Preprocesamiento de datos. Preparación
Minería de Datos: Preprocesamiento y clasificación
Máster en Ciencias de Datos e Ingeniería de Computadores
Repaso: Manejo de Pandas
Selección de datos
Antes de nada vamos a recordar cómo seleccionar atributos e instancias.
Cargamos pandas.
Leemos datos
Podemos consultar los atributos con:
Selección de atributos
Sobre este conjunto de datos haremos las siguientes operaciones de selección (en todas ellas el resultado es un nuevo conjunto de datos):
- selección de atributos concretos.
- selección de instancias concretas.
- selección de 3 variables en concreto: name, height, gender.
- todas las variables excepto las indicadas a continuación: birth_year y gender.
Selección de instancias
Es fácil filtrar un valor numérico o por valor exacto:
Se combina con & y | (no dobles) usando paréntesis:
Es más difícil si queremos filtrar según uno o varios valores:
Por varios valores
| name | height | mass | hair_color | skin_color | eye_color | homeworld | species | |
|---|---|---|---|---|---|---|---|---|
| 28 | Wicket Systri Warrick | 88.0 | 20 | brown | brown | brown | Endor | Ewok |
| 50 | Eeth Koth | 171.0 | NaN | black | brown | brown | Iridonia | Zabrak |
| 67 | Dexter Jettster | 198.0 | 102 | none | brown | yellow | Ojom | Besalisk |
| 77 | Tarfful | 234.0 | 136 | brown | brown | blue | Kashyyyk | Wookiee |
Expresiones regulares
| name | height | mass | hair_color | skin_color | eye_color | homeworld | species | |
|---|---|---|---|---|---|---|---|---|
| 2 | R2-D2 | 96.0 | 32 | NaN | white, blue | red | Naboo | Droid |
| 37 | Watto | 137.0 | NaN | black | blue, grey | yellow | Toydaria | Toydarian |
| 43 | Ayla Secura | 178.0 | 55 | none | blue | hazel | Ryloth | Twi'lek |
| 44 | Dud Bolt | 94.0 | 45 | none | blue, grey | yellow | Vulpter | Vulptereen |
| 45 | Gasgano | 122.0 | NaN | none | white, blue | black | Troiken | Xexto |
| 55 | Mas Amedda | 196.0 | NaN | none | blue | blue | Champala | Chagrian |
| 71 | Ratts Tyerell | 79.0 | 15 | none | grey, blue | NaN | Aleen Minor | Aleena |
| 75 | Shaak Ti | 178.0 | 57 | none | red, blue, white | black | Shili | Togruta |
Renombrado de variables
Se puede hacer directamente editando columns:
Index(['V1', 'V2', 'V3', 'V4', 'V5', 'V6', 'V7', 'V8'], dtype='object')Pero lo suyo es renombrar usando un diccionario:
Conocer los tipos originales del dataset
info nos devuelve los tipos (object son string)
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 87 entries, 0 to 86
Data columns (total 10 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 name 87 non-null object
1 height 81 non-null float64
2 mass 59 non-null object
3 hair_color 81 non-null object
4 skin_color 85 non-null object
5 eye_color 84 non-null object
6 birth_year 43 non-null object
7 gender 84 non-null object
8 homeworld 77 non-null object
9 species 82 non-null object
dtypes: float64(1), object(9)
memory usage: 6.9+ KBPara conocer información sobre los valores numéricos se puede hacer:
Y más en detalle se puede usar describe con un atributo:
Para ver las frecuencias se puede usar values_counts().
Human 35
Droid 5
Gungan 3
Mirialan 2
Wookiee 2
Name: species, dtype: int64Se puede normalizar (y no ordenar si se quiere):
value_counts() también permite medir frecuencia de combinaciones:
species hair_color
Human brown 0.181818
black 0.103896
none 0.038961
blond 0.038961
Gungan none 0.038961
Twi'lek none 0.025974
Mirialan black 0.025974
Human white 0.025974
dtype: float64Valores perdidos
Valores perdidos
Un problema habitual suele consistir en la presencia de datos datos.
Es importante tener claro cómo leer los datos indicando la posible ausencia de valor, usando na_values:
Hay múltiples técnicas para tratar los datos perdidos. Es importante valorar si la técnica de aprendizaje es capaz de trabajar con datos perdidos o no.
Para conocer los nulos (en porcentaje):
Opción directa (eliminar nulos)
Se pueden eliminar o bien los atributos que tienen demasiados nulos, o eliminar tuplas.
Eliminar atributos que superen un umbral:
Eliminar todas las filas con algún nulo
nombre edad color_pelo ciudad
3 Virginia 41.0 negro ParisTratar valores perdidos con paquetes externos
Dado que el aprendizaje en scikit-learn no es compatible con valores perdidos, vamos a probar distintas opciones que la propia librería nos permite.
Para probar los métodos añadidos nulos al dataset:
#Prepare the dataset to test sk-learn imputation values tools
np.random.seed(42)
rows = np.random.randint(0, np.shape(X_iris)[0], 50)
# No modifico la última característica
cols = np.random.randint(0, np.shape(X_iris)[1]-1, 50)
X_iris_missing = X_iris.to_numpy()
#Add missing values in random entries from the iris dataset
X_iris_missing[rows, cols] = np.NaN
X_iris_missing = pd.DataFrame(X_iris_missing, columns=X_iris.columns)Tenemos ahora nulos
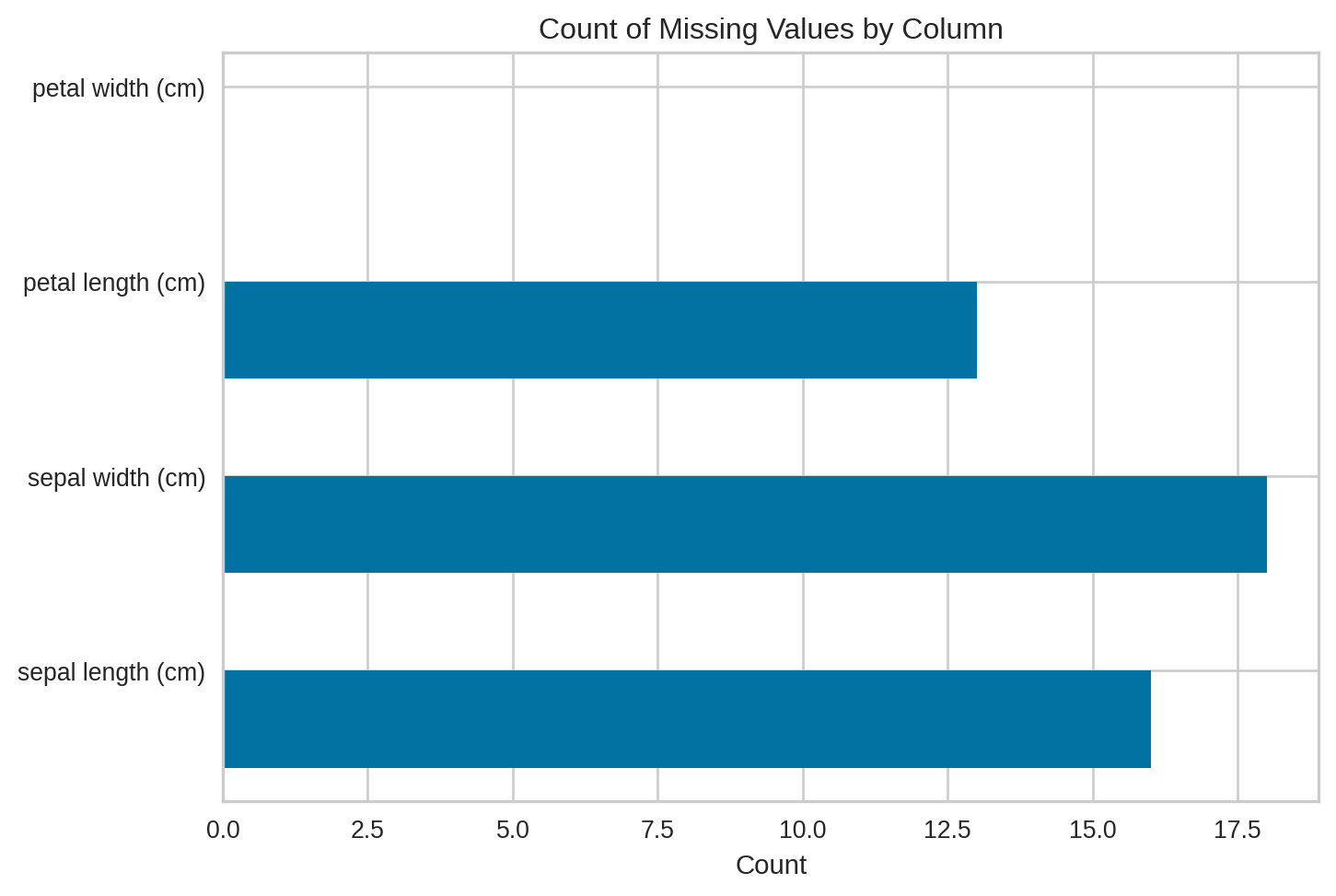
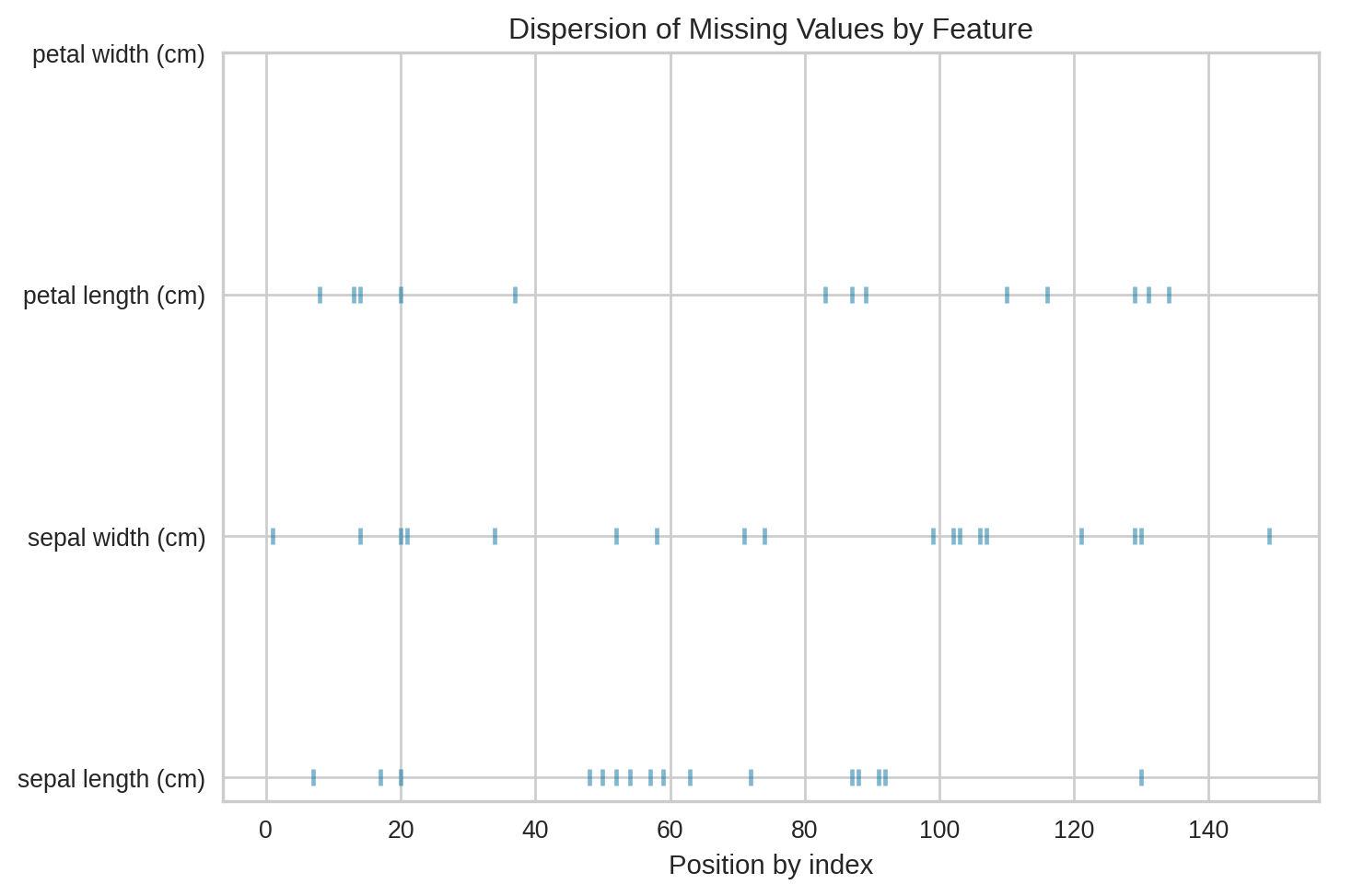
Visualizando perdidos
El paquete yellowbricks presenta muchas opciones visuales.
Imputación Univariante
Los objetos de tipo Impute permite reemplazar los valores nulos. Para ello pueden usar un valor constante o una estadística (media, mediana o más frecuente) para cada columna con nulos.
from sklearn.impute import SimpleImputer
# strategy puede ser "mean", "median", "most_frequent", "constant".
imp = SimpleImputer(missing_values=np.nan, strategy='mean')
imputed_X = pd.DataFrame(imp.fit_transform(X_iris_missing), columns=X_iris.columns)
print(imputed_X.iloc[:10,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.100000 3.500000 1.400000 0.2
1 4.900000 3.060606 1.400000 0.2
2 4.700000 3.200000 1.300000 0.2
3 4.600000 3.100000 1.500000 0.2
4 5.000000 3.600000 1.400000 0.2
5 5.400000 3.900000 1.700000 0.4
6 4.600000 3.400000 1.400000 0.3
7 5.832836 3.400000 1.500000 0.2
8 4.400000 2.900000 3.759124 0.2
9 4.900000 3.100000 1.500000 0.1Imputación Multivariante
En este caso cara característica con valores perdidos se modela en función de otras usadas para estimar la imputación.
from sklearn.experimental import enable_iterative_imputer
#Build an iterative imutation object and fit it to the data
from sklearn.impute import IterativeImputer
imp = IterativeImputer(max_iter=10, random_state=0).fit(X_iris_missing)
imputed_X = pd.DataFrame(imp.transform(X_iris_missing), columns=X_iris.columns)
print(imputed_X.iloc[:10,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.100000 3.500000 1.40000 0.2
1 4.900000 3.319683 1.40000 0.2
2 4.700000 3.200000 1.30000 0.2
3 4.600000 3.100000 1.50000 0.2
4 5.000000 3.600000 1.40000 0.2
5 5.400000 3.900000 1.70000 0.4
6 4.600000 3.400000 1.40000 0.3
7 5.009993 3.400000 1.50000 0.2
8 4.400000 2.900000 1.35012 0.2
9 4.900000 3.100000 1.50000 0.1Usando KNN
Se pueden imputar usando el algoritmo de K vecinos (KNN). Para cada atributo perdido se calcula a partir de los K vecinos más cercanos que no sea nulo. Los vecinos pueden ser diferentes para cada atributo.
Si no encuentra vecinos sin nulos, el atributo es borrado.
from sklearn.impute import KNNImputer
Knn_imp = KNNImputer(n_neighbors=4).fit(X_iris_missing)
imputed_X = pd.DataFrame(Knn_imp.transform(X_iris_missing), columns=X_iris.columns)
print(imputed_X.iloc[:10,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.10 3.50 1.400 0.2
1 4.90 3.45 1.400 0.2
2 4.70 3.20 1.300 0.2
3 4.60 3.10 1.500 0.2
4 5.00 3.60 1.400 0.2
5 5.40 3.90 1.700 0.4
6 4.60 3.40 1.400 0.3
7 5.15 3.40 1.500 0.2
8 4.40 2.90 1.375 0.2
9 4.90 3.10 1.500 0.1Visualizando reparto de nulos
También se puede ver la distribución de nulos en las instancias (por ver si hay instancias con muchos concentrados).
Sklearn permite mostrar visualmente los valores nulos con MissingIndicator.
from sklearn.impute import MissingIndicator
indicator = MissingIndicator(missing_values=np.nan).fit(X_iris_missing)
print(indicator.transform(X_iris_missing)[:8,:])
print(indicator.features_)[[False False False]
[False True False]
[False False False]
[False False False]
[False False False]
[False False False]
[False False False]
[ True False False]]
[0 1 2]Visualmente se puede mostrar usando yellobrick
La otra opción es cuando se aplique el modelo, añadir el add_indicator (falso por defecto) para que lo muestre.
#Build an Knn imutation object and fit it to the data, setting add_indicator=True
Knn_imp = KNNImputer(n_neighbors=4, add_indicator=True).fit(X_iris_missing)
print(X_iris_missing.head(5))
imputed_X = pd.DataFrame(Knn_imp.transform(X_iris_missing))
print(imputed_X.head(5)) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 NaN 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
0 1 2 3 4 5 6
0 5.1 3.50 1.4 0.2 0.0 0.0 0.0
1 4.9 3.45 1.4 0.2 0.0 1.0 0.0
2 4.7 3.20 1.3 0.2 0.0 0.0 0.0
3 4.6 3.10 1.5 0.2 0.0 0.0 0.0
4 5.0 3.60 1.4 0.2 0.0 0.0 0.0Normalización de entrada
Estandarización
Estandarización es un requisito de muchos modelos de ML, como los basados en distancias.
scikit-learn permite hacer estandarización, hay múltiples opciones
#Build a preprocessing object
from sklearn.preprocessing import StandardScaler
iris_dataset = datasets.load_iris(as_frame=True)
X_iris = iris_dataset.data.copy()
scaler = StandardScaler().fit(X_iris)
#Check the mean and the std of the training set
print(scaler.mean_)
print(scaler.scale_)[5.84333333 3.05733333 3.758 1.19933333]
[0.82530129 0.43441097 1.75940407 0.75969263]Una vez entrenado se puede aplicar:
X_iris_scaled = scaler.transform(X_iris)
print(X_iris.iloc[:5,:])
print("StandardScaler: ")
print(X_iris_scaled[:5,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
StandardScaler:
[[-0.90068117 1.01900435 -1.34022653 -1.3154443 ]
[-1.14301691 -0.13197948 -1.34022653 -1.3154443 ]
[-1.38535265 0.32841405 -1.39706395 -1.3154443 ]
[-1.50652052 0.09821729 -1.2833891 -1.3154443 ]
[-1.02184904 1.24920112 -1.34022653 -1.3154443 ]]Confirmemos:
#Transform the dataset using the preprocessin object and check results
X_scaled = pd.DataFrame(scaler.fit_transform(X_iris), columns=X_iris.columns)
print(X_scaled.mean(axis=0))
print(X_scaled.std(axis=0))sepal length (cm) -4.736952e-16
sepal width (cm) -7.815970e-16
petal length (cm) -4.263256e-16
petal width (cm) -4.736952e-16
dtype: float64
sepal length (cm) 1.00335
sepal width (cm) 1.00335
petal length (cm) 1.00335
petal width (cm) 1.00335
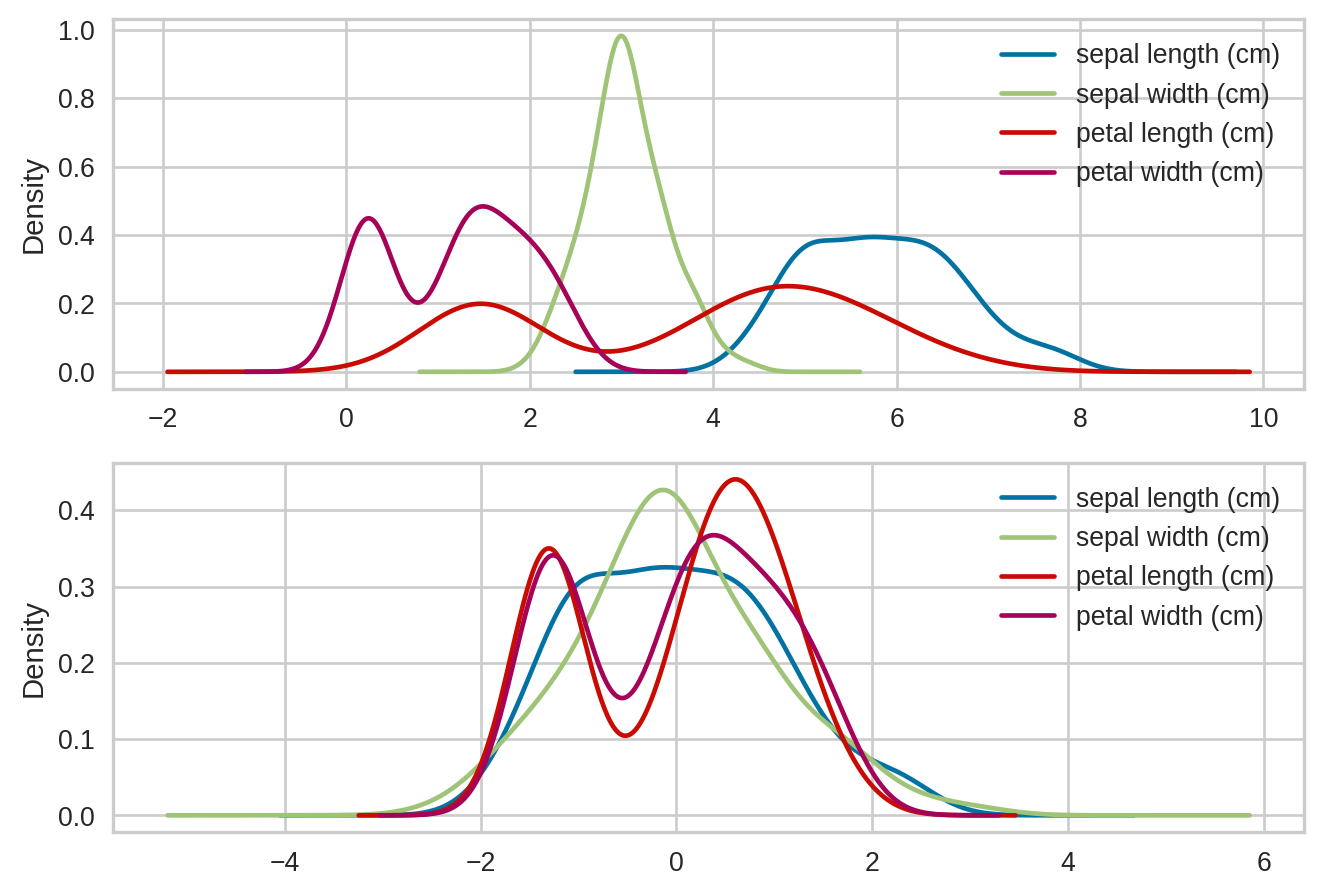
dtype: float64Visualmente
Otro muy común es MinMaxScaler:
from sklearn.preprocessing import MinMaxScaler
X_iris_scaled2 = MinMaxScaler().fit_transform(X_iris)
print(X_iris.iloc[:5,:])
print("MinMaxScaler: ")
print(X_iris_scaled2[:5,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
MinMaxScaler:
[[0.22222222 0.625 0.06779661 0.04166667]
[0.16666667 0.41666667 0.06779661 0.04166667]
[0.11111111 0.5 0.05084746 0.04166667]
[0.08333333 0.45833333 0.08474576 0.04166667]
[0.19444444 0.66666667 0.06779661 0.04166667]]Normalización
La normalización es escalar las muestras individuales para que tenga una normal unidad.
Es esencial para espresiones cuadráticas, o que usen un kernel que mida similaridad de pares de instancias.
from sklearn.preprocessing import normalize
print(X_iris.iloc[:4,:])
X_normalized = pd.DataFrame(normalize(X_iris), columns=X_iris.columns)
print(X_normalized.iloc[:4,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 0.803773 0.551609 0.220644 0.031521
1 0.828133 0.507020 0.236609 0.033801
2 0.805333 0.548312 0.222752 0.034269
3 0.800030 0.539151 0.260879 0.034784Atributos categóricos
Codificando atributos categóricos
Es común atributos con valores categóricos. Scikit-learn no es capaz de procesarlos, por lo que es necesario transformarlo a valores numéricos.
LabelEncoder y OrdinalEncoder: Asigna un valor numérico por cada categoría.
OneHotEncoder: Codifica cada categoría usando una nueva columna.
Los Label/OrdinalEncoder asigna un orden entre las categorías que suele ser ‘falso’ si ese concepto no existe.
LabelEncoder y OrdinalEncoder
Ambos asignan un valor numérico distinto a cada categoría.
Diferencia:
OrdinalEncoderpuede procesar varias columnas, se usa para características.LabelEncodersolo procesa un elemento, se usa para el atributo objetivo (target).
Advertencia: Evitar hacer esto:
from sklearn.preprocessing import LabelEncoder
targets_train = ["rubio", "moreno", "pelirrojo", "azul"]
targets_test = ["moreno", "pelirrojo"]
targets_train_num = LabelEncoder().fit_transform(targets_train)
targets_test_num = LabelEncoder().fit_transform(targets_test)
print(targets_train)
print(targets_train_num)
print(targets_test)
print(targets_test_num)['rubio', 'moreno', 'pelirrojo', 'azul']
[3 1 2 0]
['moreno', 'pelirrojo']
[0 1]Las etiquetas no coinciden.
Para evitarlo hay que hacer fit solo con el de entrenamiento.
labeler_target = LabelEncoder()
targets_train_num = labeler_target.fit_transform(targets_train)
targets_test_num = labeler_target.transform(targets_test)
print(targets_train)
print(targets_train_num)
print(targets_test)
print(targets_test_num)['rubio', 'moreno', 'pelirrojo', 'azul']
[3 1 2 0]
['moreno', 'pelirrojo']
[1 2]Guardar siempre los labeler (diccionario por nombre, …).
Ejemplo:
data_train_df = pd.DataFrame({'age': [30, 41, 42, 21],
'pelo': targets_train,
'ojos': ['azules', 'verdes', 'marrones', 'marrones']})
data_test_df = pd.DataFrame({'age': [25, 23],
'pelo': targets_test,
'ojos': ['verdes', 'azules']})
print(data_train_df) age pelo ojos
0 30 rubio azules
1 41 moreno verdes
2 42 pelirrojo marrones
3 21 azul marronesVamos a aplicar el etiquetado.
- Opción 1: Sólo con Label Encoder:
labelers = {}
cols = {}
atribs = ["pelo", "ojos"]
data_train_num = data_train_df.copy()
data_test_num = data_test_df.copy()
for i in atribs:
cols[i] = LabelEncoder()
data_train_num[i] = cols[i].fit_transform(data_train_num[i])
data_test_num[i] = cols[i].transform(data_test_num[i])
print(data_train_num)
print(data_test_num) age pelo ojos
0 30 3 0
1 41 1 2
2 42 2 1
3 21 0 1
age pelo ojos
0 25 1 2
1 23 2 0- Opción 2: Usando
OrdinalEncoder
from sklearn.preprocessing import OrdinalEncoder
atribs = ["pelo", "ojos"]
labelers = OrdinalEncoder(dtype=np.int32) # Por defecto usa float
data_train_num = data_train_df.copy()
data_test_num = data_test_df.copy()
data_train_num[atribs] = labelers.fit_transform(data_train_df[atribs])
data_test_num[atribs] = labelers.transform(data_test_df[atribs])
print(data_train_num)
print(data_test_num) age pelo ojos
0 30 3 0
1 41 1 2
2 42 2 1
3 21 0 1
age pelo ojos
0 25 1 2
1 23 2 0Inversión
También se puede invertir el etiquetado:
LabelEncoder, OrdinalEncoder y orden
Esta codificación está considerando un orden entre categorías.
En algunos casos como [‘pequeño’, ‘mediano’, ‘grande’] puede tener sentido pero la mayoría de las veces no.
Cuando no (como ‘pelo’ o ‘color’ del ejemplo anterior) es necesario aplicar OneHotEncoder.
OneHotEncoder crea una columna por categoría (ej: ‘azul’) indicando si se cumple o no.
Aumenta el número de columnas.
Evita suponer un orden.
Se puede convertir a dataframe:
new_columns = encoder.get_feature_names_out()
print(new_columns)
data_train_hot = pd.DataFrame(data_train_hot, columns=new_columns)
# Copio el resto de atributos
data_train_hot['age'] = data_train_df['age']
print(data_train_hot)['pelo_azul' 'pelo_moreno' 'pelo_pelirrojo' 'pelo_rubio' 'ojos_azules'
'ojos_marrones' 'ojos_verdes']
pelo_azul pelo_moreno pelo_pelirrojo pelo_rubio ojos_azules \
0 0 0 0 1 1
1 0 1 0 0 0
2 0 0 1 0 0
3 1 0 0 0 0
ojos_marrones ojos_verdes age
0 0 0 30
1 0 1 41
2 1 0 42
3 1 0 21 Dummies en Pandas
Pandas ya soporta el hotencoding, pero presenta problemas.
| pelo_azul | pelo_moreno | pelo_pelirrojo | pelo_rubio | ojos_azules | ojos_marrones | ojos_verdes | |
|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 |
| 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 |
| 2 | 0 | 0 | 1 | 0 | 0 | 1 | 0 |
| 3 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
Recomiendo usar OneHotEncoder por tener más opciones.
Valores binarios
Si el valor numérico es binario, ej: vivo/muerto no es necesario aplicar el hotencoding.
columns = ["Employed", "Place", 'Browser']
X = [['employed', 'from US', 'uses Safari'], ['unemployed', 'from Europe', 'uses Firefox'], ['unemployed', 'from Asia', 'uses Chrome']]
enc = OneHotEncoder(drop='if_binary')
trans_X = enc.fit_transform(X)
transformed_X = pd.DataFrame(trans_X.toarray(), columns=enc.get_feature_names_out())
print(transformed_X) x0_unemployed x1_from Asia x1_from Europe x1_from US x2_uses Chrome \
0 0.0 0.0 0.0 1.0 0.0
1 1.0 0.0 1.0 0.0 0.0
2 1.0 1.0 0.0 0.0 1.0
x2_uses Firefox x2_uses Safari
0 0.0 1.0
1 1.0 0.0
2 0.0 0.0 LabelBinarizer
OneHotEncoder es para convertir atributos, LabelBinarizer es para convertir el target (por ejemplo: para redes neuronales).
La diferencia principal es:
LabelBinarizerdevuelve una matriz numpy,OneHotEncoderdevuelve por defecto matriz sparse.LabelBinarizerdevuelve de tipo entero,OneHotEncoderdevuelve por defecto de tipofloat.OneHotEncoderpuede convertir distintos atributos a la vez,LabelBinarizerno.
from sklearn.preprocessing import LabelBinarizer
lb = LabelBinarizer()
lb.fit_transform(data_train_df['ojos'])
# Da error
# lb.fit_transform(data_train_df['ojos','pelo'])array([[1, 0, 0],
[0, 0, 1],
[0, 1, 0],
[0, 1, 0]])¿Cuándo usar LabelBinarizer?
Para etiquetar el objetivo.
Multilabel
¿Qué pasa cuando un atributo es una combinación de valores?
Ejemplo: género de una película.
La clase Multilabel permite identificar:
Aviso: Trabaja con vector de conjunto, no con vector:
mlb = MultiLabelBinarizer()
print(mlb.fit_transform(['sci-fi', 'thriller', 'comedy']))
list(mlb.classes_)[[1 1 0 0 1 0 1 0 0 0 0 1 0 0]
[0 0 0 1 0 1 1 1 0 0 1 0 1 0]
[0 1 1 1 0 0 0 0 1 1 0 0 0 1]]['-', 'c', 'd', 'e', 'f', 'h', 'i', 'l', 'm', 'o', 'r', 's', 't', 'y']La otra opción es un vector de vectores:
Pipelines y ColumnTransformer
Aplicar OneHotEncoder
Scikit-learn permite combinar transformaciones con ColumnTransformer.
from sklearn.compose import make_column_transformer
transformer = make_column_transformer(
(OneHotEncoder(), ['pelo', 'ojos']),
remainder='passthrough') # Para ignorar el resto y no dar error
transformed = transformer.fit_transform(data_train_df)
data_train_num = pd.DataFrame(transformed, columns=transformer.get_feature_names_out())
print(data_train_num) onehotencoder__pelo_azul onehotencoder__pelo_moreno \
0 0.0 0.0
1 0.0 1.0
2 0.0 0.0
3 1.0 0.0
onehotencoder__pelo_pelirrojo onehotencoder__pelo_rubio \
0 0.0 1.0
1 0.0 0.0
2 1.0 0.0
3 0.0 0.0
onehotencoder__ojos_azules onehotencoder__ojos_marrones \
0 1.0 0.0
1 0.0 0.0
2 0.0 1.0
3 0.0 1.0
onehotencoder__ojos_verdes remainder__age
0 0.0 30.0
1 1.0 41.0
2 0.0 42.0
3 0.0 21.0 ColumnTransformer permite procesar distintos datasets.
En conjunción con make_column_selector (que permite filtrar atributos por su tipo) es muy potente y cómodo.
from sklearn.compose import make_column_transformer
from sklearn.compose import make_column_selector
X = pd.DataFrame({'city': ['London', 'London', 'Paris', 'Sallisaw'],
'rating': [5, 3, 4, 5]})
ct = make_column_transformer(
(StandardScaler(),
make_column_selector(dtype_include=np.number)), # rating
(OneHotEncoder(),
make_column_selector(dtype_include=object))) # city
ct.fit_transform(X)array([[ 0.90453403, 1. , 0. , 0. ],
[-1.50755672, 1. , 0. , 0. ],
[-0.30151134, 0. , 1. , 0. ],
[ 0.90453403, 0. , 0. , 1. ]])Pipelines
Facilitan aplicar distintos preprocesamientos.
Un pipeline se compone de una serie de transformaciones que van sufriendo el dataset (se puede incluir el modelo a aprender).
Un pipeline se usa igual que un modelo.
from sklearn.svm import SVC
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import Pipeline
iris_targets = iris_dataset.target
from sklearn.model_selection import cross_val_score, train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_iris, iris_targets,
random_state=0)
pipe = Pipeline([('scaler', StandardScaler()), ('svc', SVC())])
pipe.fit(X_train, y_train)
pipe.score(X_test, y_test)0.9736842105263158También se puede aplicar con validación cruzada:
from sklearn.model_selection import cross_val_score
pipe = Pipeline([('scaler', StandardScaler()), ('svc', SVC())])
scores = cross_val_score(pipe, X_iris, iris_targets, cv=5)
print(scores)
print(scores.mean())[0.96666667 0.96666667 0.96666667 0.93333333 1. ]
0.9666666666666666Por comodidad se puede usar make_pipeline con tantos atributos como procesamientos y/o mdelos.
Pipelines y ColumnTransformer
También se pueden combinar con ColumnTransformer.
from sklearn.model_selection import cross_val_score
trans = make_column_transformer(
(StandardScaler(), ["age"]),
(OneHotEncoder(), ["pelo", "ojos"])
)
trans.fit_transform(data_train_df)
pipe = make_pipeline(trans, SVC())
print(data_train_df)
pipe.fit(data_train_df, [0, 0, 1, 1])
print(data_test_df)
pipe.predict(data_test_df) age pelo ojos
0 30 rubio azules
1 41 moreno verdes
2 42 pelirrojo marrones
3 21 azul marrones
age pelo ojos
0 25 moreno verdes
1 23 pelirrojo azulesarray([0, 0])Discretización
Discretizando usando umbral
A veces no nos interesa mostrar si un valor numérico es suficientemente alto o no. Vienen bien para algunos clasificadores, como el Bernoulli Restricted Boltzmann Machine, y son muy populares en procesamiento de texto.
Por ejemplo, a partir del número de cigarros al día identificar si es un fumador habitual.
Discretización usando rangos
A menudo no nos interesa un valor numéricos (ej: age) sino convertirlo en un conjunto discreto de valores (joven, adulto, mayor).
La clase K-bins permite discretizar.
from sklearn.preprocessing import KBinsDiscretizer
#Build a discretizer object indicating three bins for every feature
est = KBinsDiscretizer(n_bins=[3, 3, 3, 3], encode='ordinal').fit(X_iris)
#Check feature maximum and minimum values
# print(np.max(X_iris, axis = 0))
# print(np.min(X_iris, axis = 0))
#Check binning intervals
print(est.bin_edges_)[array([4.3, 5.4, 6.3, 7.9]) array([2. , 2.9, 3.2, 4.4])
array([1. , 2.63333333, 4.9 , 6.9 ])
array([0.1 , 0.86666667, 1.6 , 2.5 ])]#Print discretization results
print(X_iris.iloc[:5,])
discretized_X = pd.DataFrame(est.transform(X_iris), columns=X_iris.columns)
print(discretized_X.iloc[:5,]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 0.0 2.0 0.0 0.0
1 0.0 1.0 0.0 0.0
2 0.0 2.0 0.0 0.0
3 0.0 1.0 0.0 0.0
4 0.0 2.0 0.0 0.0Distintas estrategias de discretización
El criterio de discretización puede ser cambiado con el parámetro strategy.
Una tendencia común sería una uniforme:
est = KBinsDiscretizer(n_bins=5, encode='ordinal', strategy='uniform')
age_disc = est.fit_transform(data_train_df[['age']])
print(est.bin_edges_)
print(age_disc)[array([21. , 25.2, 29.4, 33.6, 37.8, 42. ])]
[[2.]
[4.]
[4.]
[0.]]No todos los rangos tienen interés, pueden concentrarse.
A menudo la mejor estrategia depende de la frecuencia (comportamiento por defecto).
est = KBinsDiscretizer(n_bins=5, encode='ordinal', strategy='quantile')
age_disc = est.fit_transform(data_train_df[['age']])
print(est.bin_edges_)
print(age_disc)[array([21. , 26.4, 32.2, 38.8, 41.4, 42. ])]
[[1.]
[3.]
[4.]
[0.]]De esta manera, discretiza más en detalle los intervalos más comunes.
La otra opción es la estrategia kmean que aplica una clasificación kmeans sobre cada algoritmo.
#Build a discretizer object indicating three bins for every feature and using the kmeans strategy
est = KBinsDiscretizer(n_bins=[3, 3, 3, 3], encode='ordinal', strategy='kmeans').fit(X_iris)
#Check binning intervals and results
print(est.bin_edges_)
discretized_X = pd.DataFrame(est.transform(X_iris), columns=X_iris.columns)
print(discretized_X.iloc[:5,])[array([4.3 , 5.53309253, 6.54877049, 7.9 ])
array([2. , 2.85216858, 3.43561538, 4.4 ])
array([1. , 2.87637037, 4.95950081, 6.9 ])
array([0.1 , 0.79151852, 1.70547504, 2.5 ])]
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 0.0 2.0 0.0 0.0
1 0.0 1.0 0.0 0.0
2 0.0 1.0 0.0 0.0
3 0.0 1.0 0.0 0.0
4 0.0 2.0 0.0 0.0Discretización usando CAIM
CAIM es un algoritmo de discretización muy usado. En Python está disponible en el paquete caimcaim:
from caimcaim import CAIMD
caim_dis = CAIMD()
caim_dis.fit(X_scaled, iris_targets)
print(X_scaled.iloc[:5,])
discretized_X = caim_dis.transform(X_scaled)
print(discretized_X.iloc[:5,:])Categorical []# 0 GLOBAL CAIM 26.636271740334553# 1 GLOBAL CAIM 17.382507167267576
# 2 GLOBAL CAIM 45.55892255892255
# 3 GLOBAL CAIM 46.16156736446592
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 -0.900681 1.019004 -1.340227 -1.315444
1 -1.143017 -0.131979 -1.340227 -1.315444
2 -1.385353 0.328414 -1.397064 -1.315444
3 -1.506521 0.098217 -1.283389 -1.315444
4 -1.021849 1.249201 -1.340227 -1.315444 sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 0.0 2.0 0.0 0.0
1 0.0 2.0 0.0 0.0
2 0.0 2.0 0.0 0.0
3 0.0 2.0 0.0 0.0
4 0.0 2.0 0.0 0.0Generación de atributos polinomiales
A veces es útil añadir complejidad a un modelo añadiendo características no lineales. scikit-learn incorpora dos estrategias:
Polinomiales.
Usando splines, trozos polinomiales.
Polinomiales
Construye atributos como combinación polinomial de los existentes.
Si la entrada es [a, b] y se usa grado 2, los atributos polinomiales serían [1, a, b, a^2, ab, b^2].
from sklearn.preprocessing import PolynomialFeatures
#Build a polynomial features generator for the squared augmentation, this transforms (X1,X2) to (1,X1,X2,X1^2,X1X2,X2^2)
poly = PolynomialFeatures(degree=2).fit(X_iris)
print(X_iris.iloc[:3,])
poly_X = pd.DataFrame(poly.transform(X_iris))
print(poly_X.iloc[:3,]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
0 1 2 3 4 5 6 7 8 9 10 11 12 \
0 1.0 5.1 3.5 1.4 0.2 26.01 17.85 7.14 1.02 12.25 4.90 0.70 1.96
1 1.0 4.9 3.0 1.4 0.2 24.01 14.70 6.86 0.98 9.00 4.20 0.60 1.96
2 1.0 4.7 3.2 1.3 0.2 22.09 15.04 6.11 0.94 10.24 4.16 0.64 1.69
13 14
0 0.28 0.04
1 0.28 0.04
2 0.26 0.04 A veces interesa solo las interacciones, se puede usar interaction_only.
#Sometimes only interaction terms are required, which can be obtained by setting interaction_only=True
poly = PolynomialFeatures(degree=2, interaction_only=True).fit(X_iris)
print(X_iris.iloc[:5,])
poly_X = pd.DataFrame(poly.transform(X_iris))
print(poly_X.iloc[:5,]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
0 1 2 3 4 5 6 7 8 9 10
0 1.0 5.1 3.5 1.4 0.2 17.85 7.14 1.02 4.90 0.70 0.28
1 1.0 4.9 3.0 1.4 0.2 14.70 6.86 0.98 4.20 0.60 0.28
2 1.0 4.7 3.2 1.3 0.2 15.04 6.11 0.94 4.16 0.64 0.26
3 1.0 4.6 3.1 1.5 0.2 14.26 6.90 0.92 4.65 0.62 0.30
4 1.0 5.0 3.6 1.4 0.2 18.00 7.00 1.00 5.04 0.72 0.28Ejercicios
Ejercicios de nulos
- Leer el dataset del fichero tanzania_water_pump_org.csv (nota: los valores perdidos están representados con cadena vacía).
Index(['id', 'amount_tsh', 'date_recorded', 'funder', 'gps_height',
'installer', 'longitude', 'latitude', 'wpt_name', 'num_private',
'basin', 'subvillage', 'region', 'region_code', 'district_code', 'lga',
'ward', 'population', 'public_meeting', 'recorded_by',
'scheme_management', 'scheme_name', 'permit', 'construction_year',
'extraction_type', 'extraction_type_group', 'extraction_type_class',
'management', 'management_group', 'payment', 'payment_type',
'water_quality', 'quality_group', 'quantity', 'quantity_group',
'source', 'source_type', 'source_class', 'waterpoint_type',
'waterpoint_type_group', 'status_group'],
dtype='object')Mostrar algún diagrama para mostrar casos de nulos.
Eliminar atributos con un número de nulos mayor que el 30%.
Mostrar distribución de nulos.
Comparar el número de filas eliminando y no eliminando nulos.
Eliminar nulos sobre el original aplicando reemplazo correspondiente.
Eliminar los nulos sobre el original aplicando el KNN.
Comparar los resultados según los pasos 5 y 6.
Ejercicios de discretización
Este ejercicio se hace sobre el IMDB “IMDB-Movie-Data.csv”.
Index(['Rank', 'Title', 'Genre', 'Description', 'Director', 'Actors', 'Year',
'Runtime (Minutes)', 'Rating', 'Votes', 'Revenue (Millions)',
'Metascore'],
dtype='object')Discretiza los ingresos en 10 intervalos de igual rango.
Discretiza los ingresos en 10 intervalos según la frecuencia.
Discretizar las películas entre populares (un 8 o más) y menos.
Ejercicios de etiquetado
Borrar la descripción.
En el dataset de IMDB etiquetar el género usando el MultiLabel.
Etiquetar el director usando etiquetado.
Etiquetar el director usando HotEncoder.
Comparar resultados entre 3 y 4.
Ejercicios de Pipeline
Aplicar con ColumnTransformer y Pipeline para automatizar las transformaciones anteriores con el IMDB (sin aplicar el paso 5 anterior).