Preprocesamiento Avanzado de datos. Reducción
Minería de Datos: Preprocesamiento y clasificación
Máster en Ciencias de Datos e Ingeniería de Computadores
Reducción de dimensionalidad
Reducción de dimensionalidad
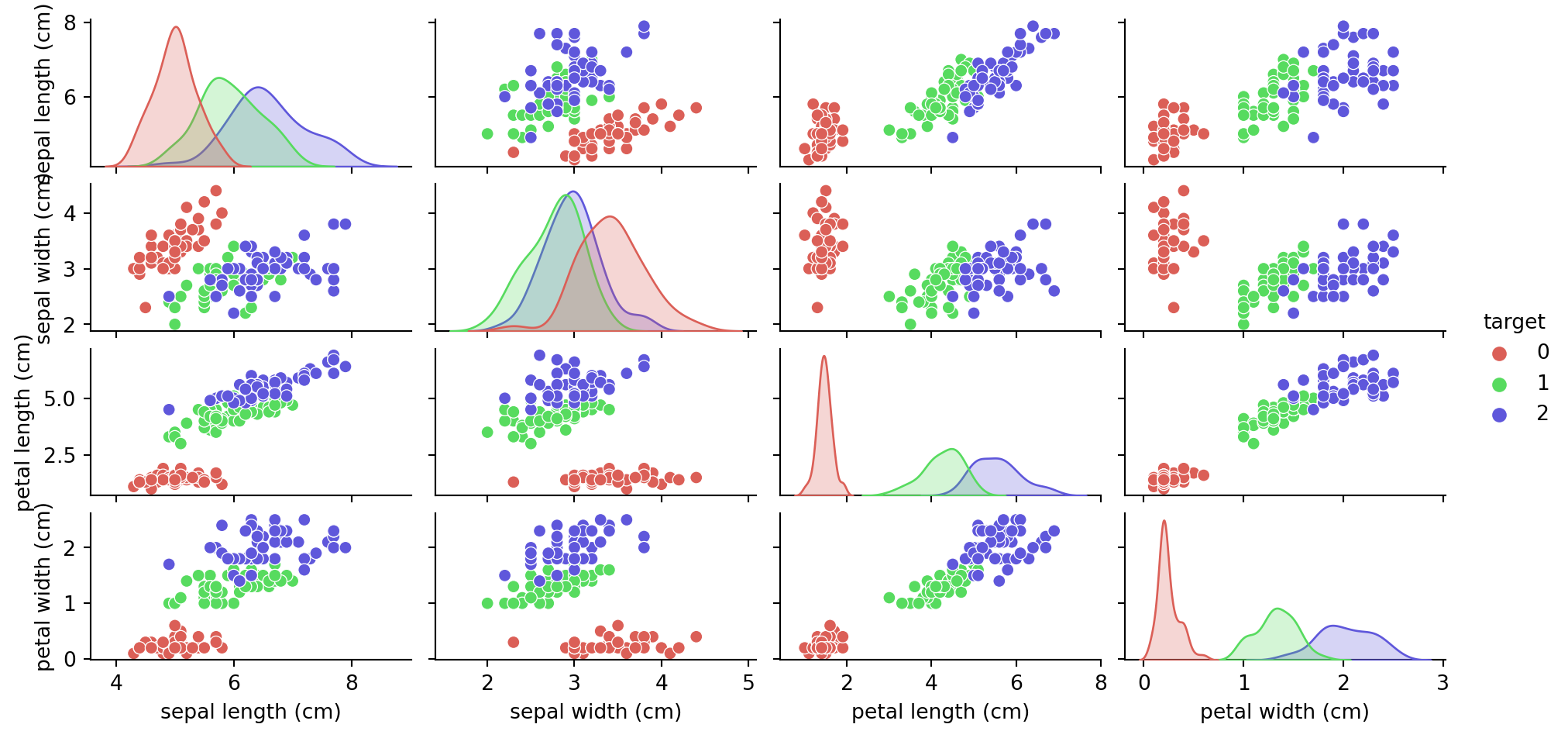
Una de las prácticas más comunes de procesamiento de datos es la reducción de la dimensional, lo cual ayuda a transformar o seleccionar las características que mejor representan la estructura, y que por tanto, son más adecuadas para el aprendizaje.
Reducción de la Dimensionalidad No supervisada
Si el número de características es alto, puede ser útil reducirlas mediante una fase no supervisada.
Principal Components Analysis (PCA)
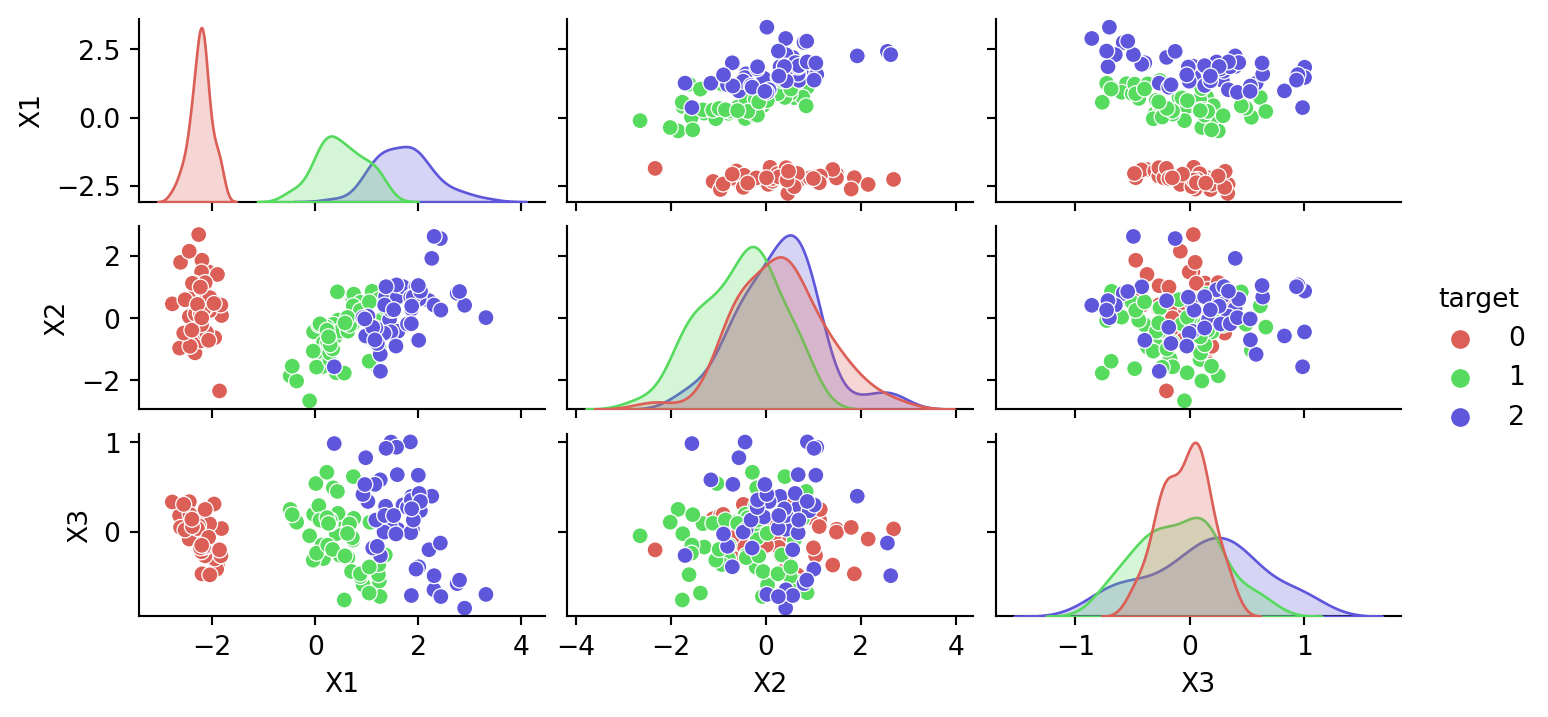
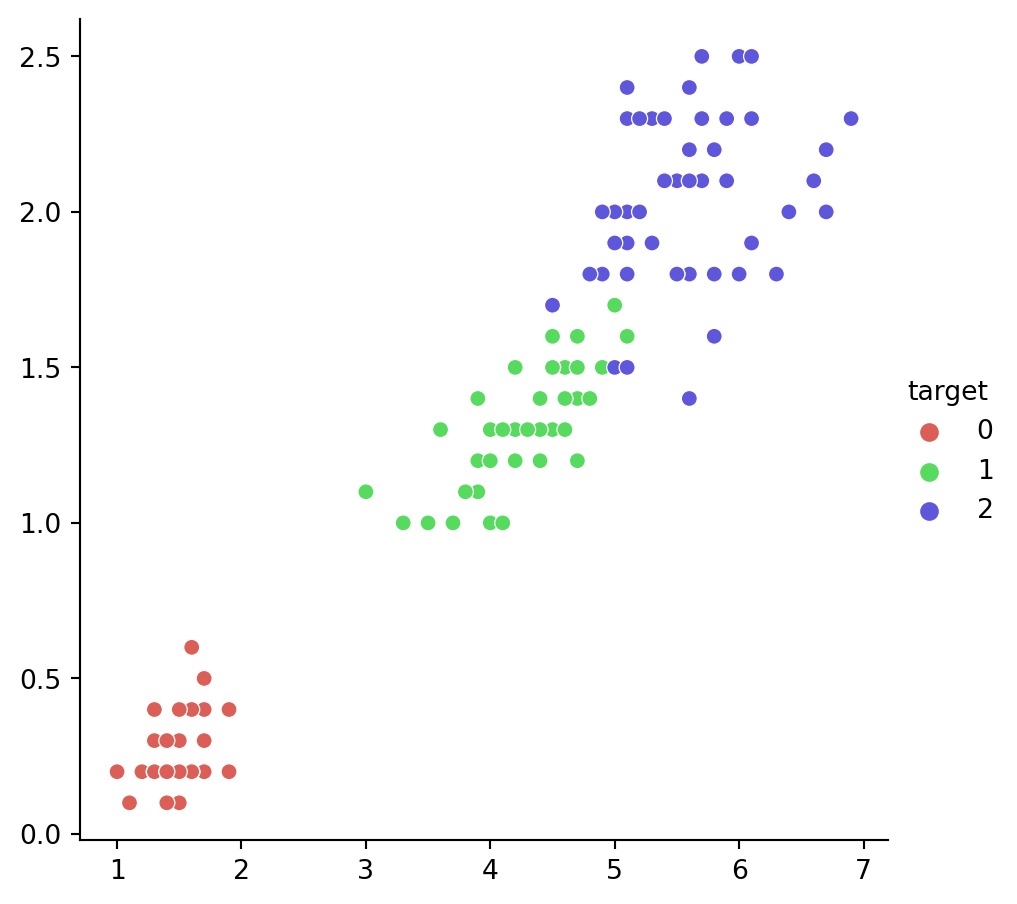
Descomponer un dataset multivariante en un conjunto de componentes ortogonales que explica la cantidad de varianza.
Aplico el PCA:
#Apply principal componentes analysis to reduce the iris number of features from 4 to 3
pca = decomposition.PCA(n_components=3)
X_reduced = pca.fit_transform(X_scaled)
print(X_scaled[:3,:])
print("Con menos dimensión")
print(X_reduced[:3,:])[[-0.90068117 1.01900435 -1.34022653 -1.3154443 ]
[-1.14301691 -0.13197948 -1.34022653 -1.3154443 ]
[-1.38535265 0.32841405 -1.39706395 -1.3154443 ]]
Con menos dimensión
[[-2.26470281 0.4800266 -0.12770602]
[-2.08096115 -0.67413356 -0.23460885]
[-2.36422905 -0.34190802 0.04420148]]Aglomeración
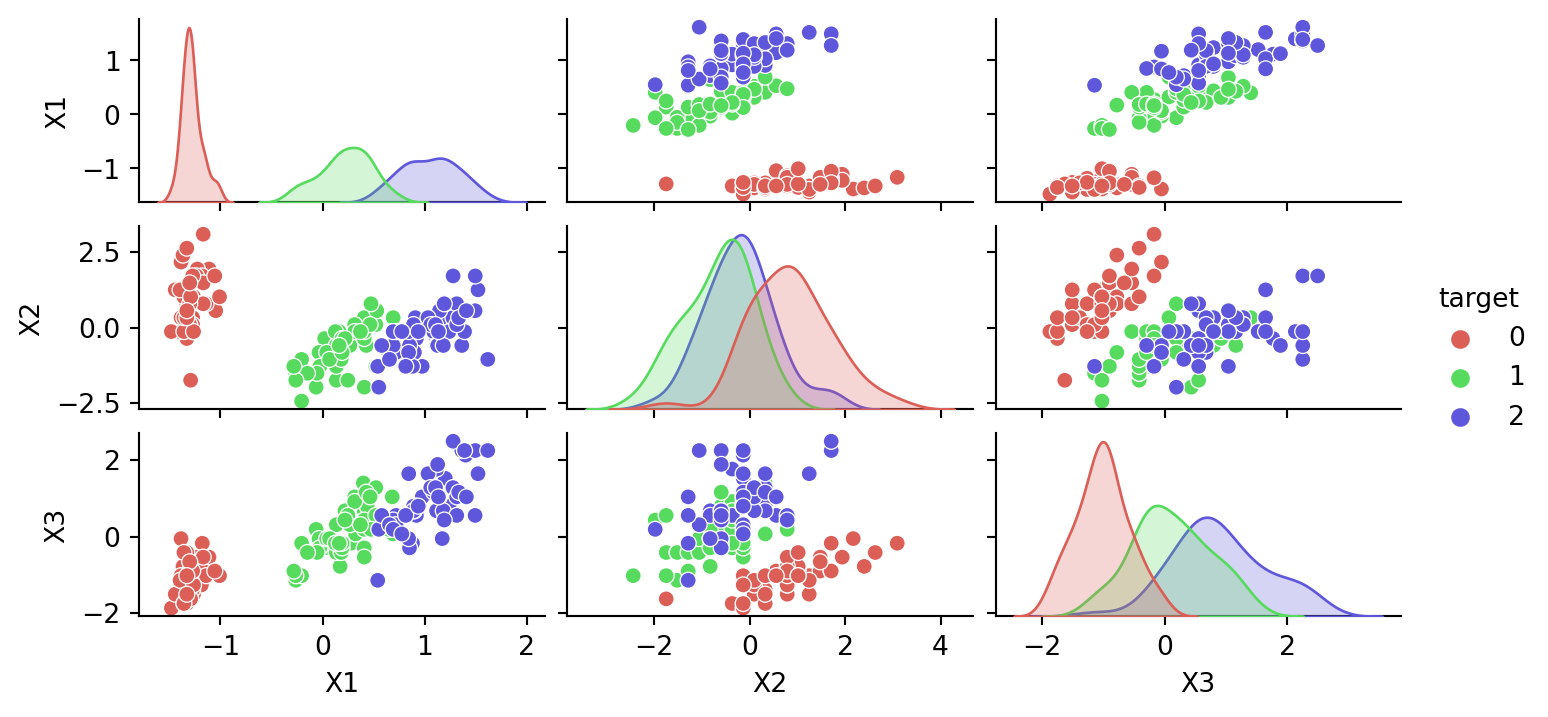
Esta técnica hace clusters de forma jerárquica y va agrupando características que se comportan de forma similar.
import numpy as np
from sklearn import cluster
#Build a FeatureAgglomeration object and transform the iris dataset for it to have 3 features
agglo = cluster.FeatureAgglomeration(n_clusters=3,
pooling_func=np.mean, linkage="ward")
X_reduced2 = agglo.fit_transform(X_scaled)
print(X_scaled[:3,:])
print("Con menos dimensión")
print(X_reduced2[:3,:])[[-0.90068117 1.01900435 -1.34022653 -1.3154443 ]
[-1.14301691 -0.13197948 -1.34022653 -1.3154443 ]
[-1.38535265 0.32841405 -1.39706395 -1.3154443 ]]
Con menos dimensión
[[-1.32783541 1.01900435 -0.90068117]
[-1.32783541 -0.13197948 -1.14301691]
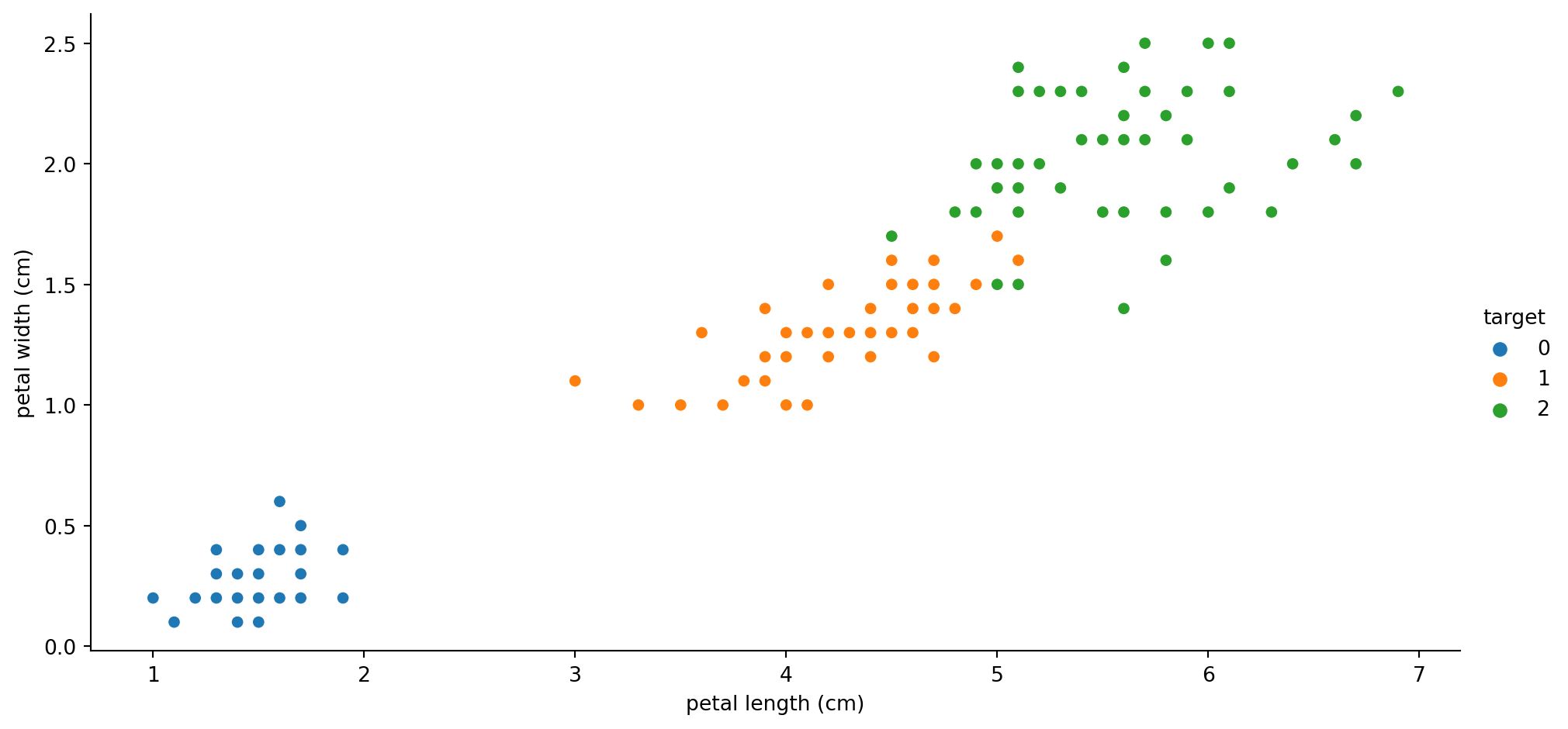
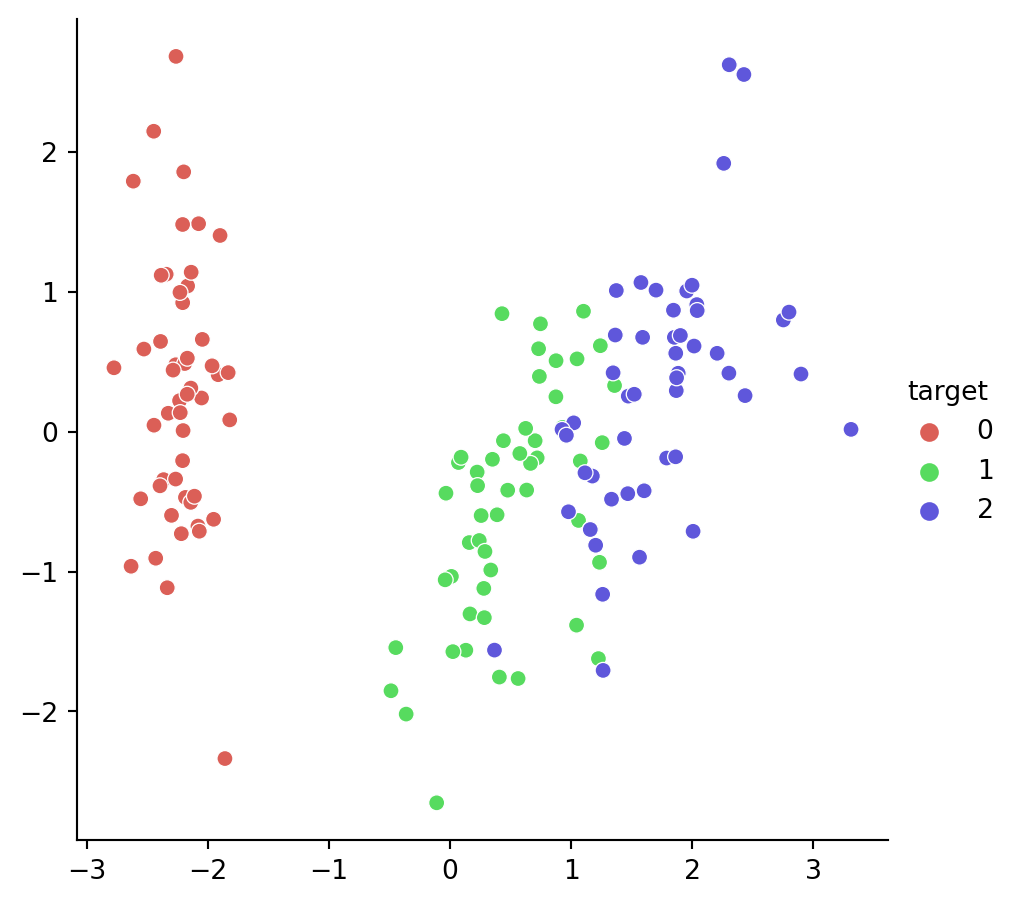
[-1.35625412 0.32841405 -1.38535265]]Visualizando en 2D el original:
Visualizando en 2D el PCA:
Visualizando en 2D la reducción mediante Aglomeración:
Selección de Características
Selección de Características
En este caso se reducen las características eligiendo las características que permitirían un mejor desempeño del clasificador.
Eliminar características con menor varianza
Es un modelo muy simple. Borra todas las características cuya varianza no cumpla un umbral.
Por defecto borra las que tengan 0 varianza.
from sklearn.feature_selection import VarianceThreshold
#Build a variance threshold selector and transform the iris dataet for it to have three features
sel = VarianceThreshold(threshold=0.2)
X_reduced3 = sel.fit_transform(X_iris)
print(X_reduced3.shape)
print(X_iris.iloc[:3,:])
print("Tras reducir")
print(X_reduced3[:3,:])(150, 3)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
Tras reducir
[[5.1 1.4 0.2]
[4.9 1.4 0.2]
[4.7 1.3 0.2]]Midiendo la correlación
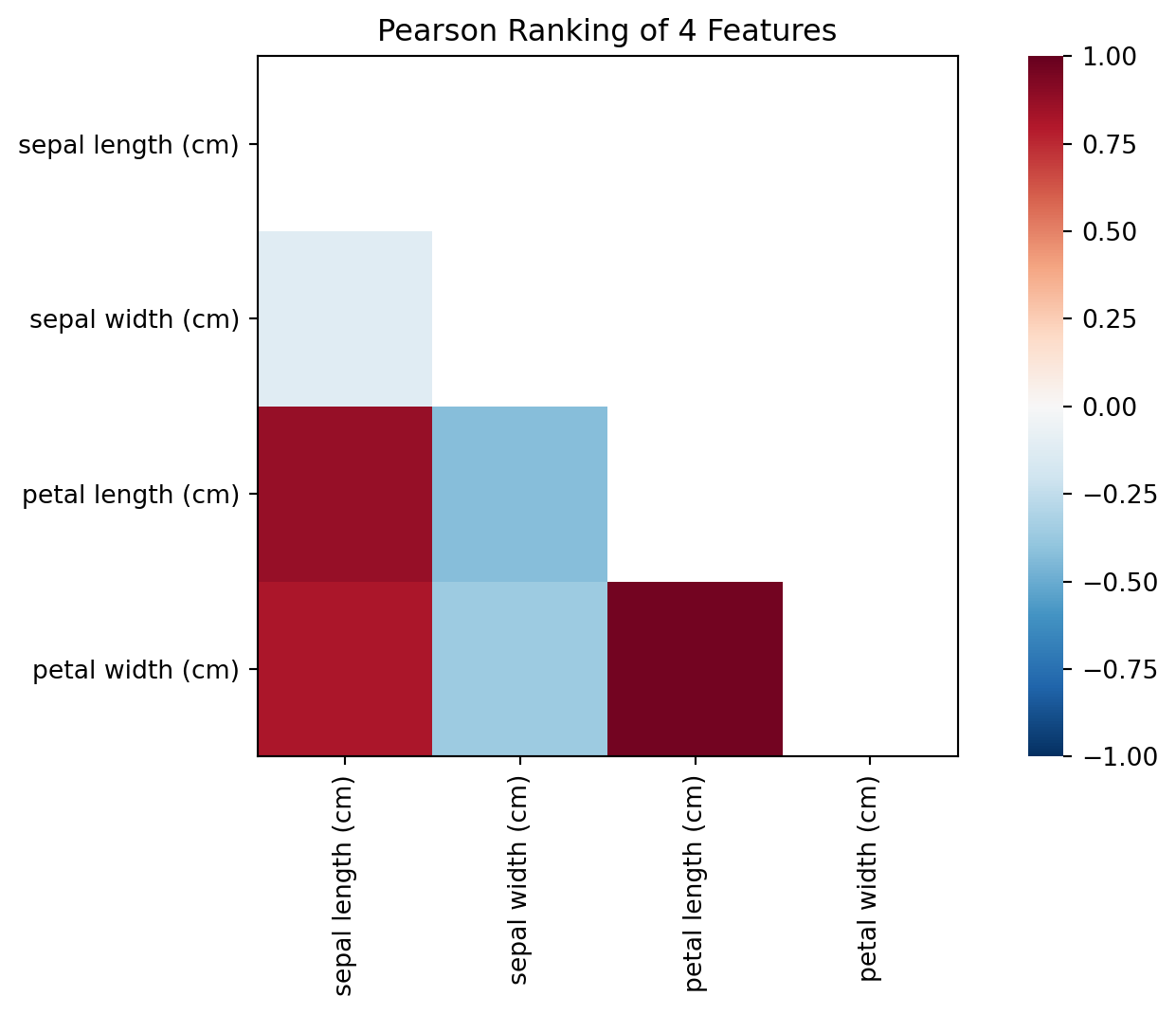
La matriz de correlación permite mostrar atributos que pueden ser redundantes:
sepal length (cm) sepal width (cm) petal length (cm) \
sepal length (cm) 1.000000 -0.117570 0.871754
sepal width (cm) -0.117570 1.000000 -0.428440
petal length (cm) 0.871754 -0.428440 1.000000
petal width (cm) 0.817941 -0.366126 0.962865
petal width (cm)
sepal length (cm) 0.817941
sepal width (cm) -0.366126
petal length (cm) 0.962865
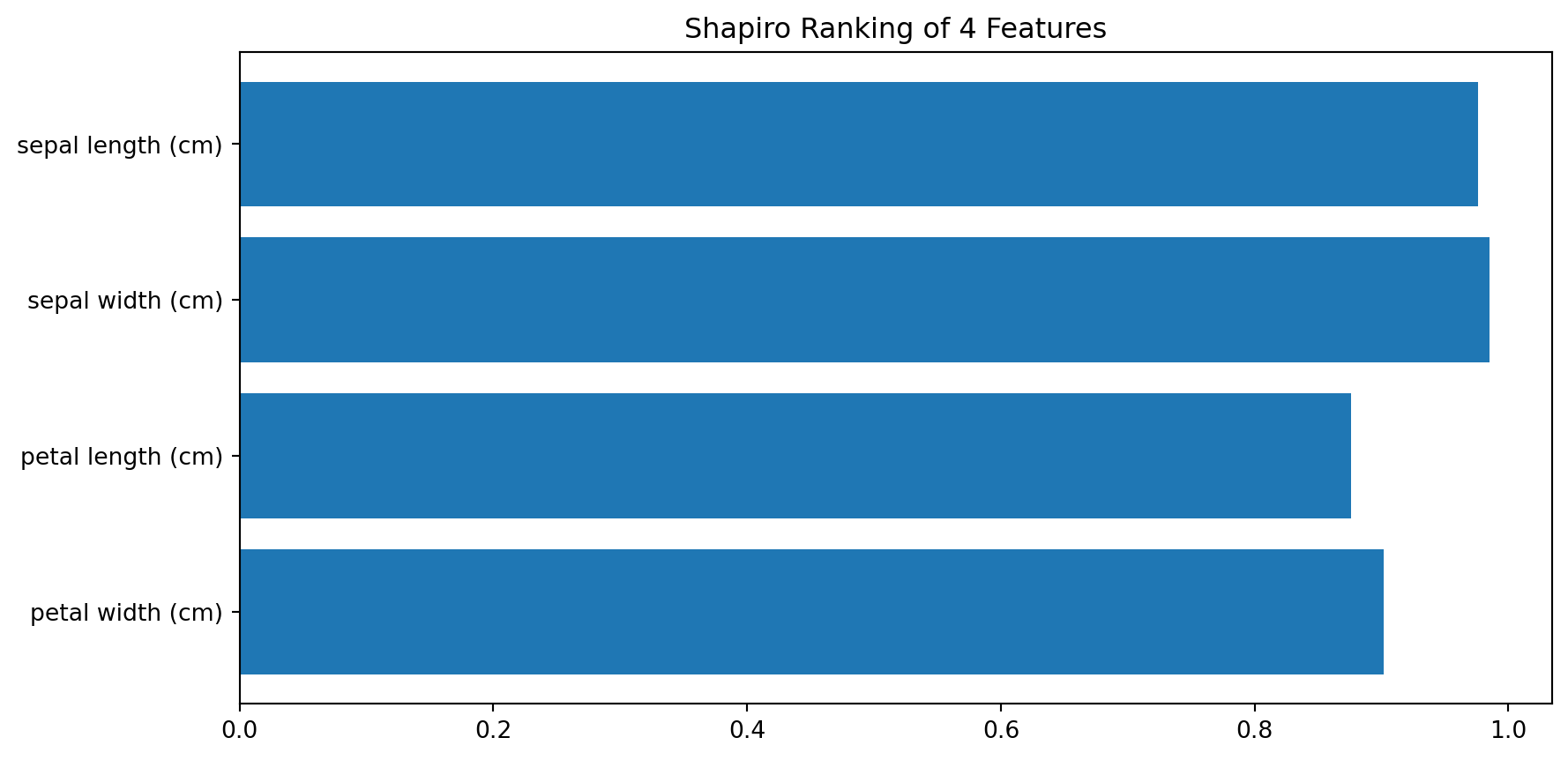
petal width (cm) 1.000000 Yellowbrick nos permite visualizarlo:
Aplicar la importancia según métrica
Selección Univariante
La selección de características univariante funciona seleccionando escogiendo las mejores características según tests univariantes.
from sklearn.feature_selection import SelectKBest, chi2, SelectPercentile
# Según el criterio chi-squared dustribution for it to have three features
sel = SelectKBest(chi2, k=3)
X_reduced4 = sel.fit_transform(X_iris, y_iris)
print(X_reduced4.shape)
print(X_iris.iloc[:3,:])
print("Tras reducir")
print(X_reduced4[:3,:])(150, 3)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
Tras reducir
[[5.1 1.4 0.2]
[4.9 1.4 0.2]
[4.7 1.3 0.2]]# Según un percentil
sel = SelectPercentile(chi2, percentile=50)
X_reduced5 = sel.fit_transform(X_iris, y_iris)
print(X_reduced5.shape)
print(X_iris.iloc[:3,:])
print("Tras reducir")
print(X_reduced5[:3,:])(150, 2)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
Tras reducir
[[1.4 0.2]
[1.4 0.2]
[1.3 0.2]]Eliminación recursiva de características
Dado un estimador que asigna pesos a características, la eliminación recursiva, recursive feature elimination (RFE) selecciona recursivamente menos y menos características.
Primero se entrena el estimador, y se mide la importancia de cada atributo.
Las menos importantes son eliminadas del conjunto de características de forma recursiva hasta que se alcanza el número de características deseadas.
Vamos a aplicarlo hasta 2 características:
from sklearn.svm import SVC
from sklearn.feature_selection import RFE
#Use a Support Vector Classifier as the base model for feature selection
svc = SVC(kernel="linear", C=1)
#Build a RFE model with the SVC to reduce the number of features of iris to 2
rfe = RFE(estimator=svc, n_features_to_select=2, step=1)
ranking = rfe.fit(X_iris, y_iris).ranking_
X_reduced_rfe = rfe.fit_transform(X_iris, y_iris)
print(ranking); print(X_reduced_rfe.shape)
print(X_iris.iloc[:3,:])
print("Reduced"); print(X_reduced_rfe[:3,:])[3 2 1 1]
(150, 2)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
Reduced
[[1.4 0.2]
[1.4 0.2]
[1.3 0.2]]Vamos a visualizarlo:
Vamos a probar otro dataset:
#Code from sklearn documentation
from sklearn.svm import SVC
from sklearn.datasets import load_digits
from sklearn.feature_selection import RFE
import matplotlib.pyplot as plt
# Load the digits dataset
digits = load_digits()
X = digits.images.reshape((len(digits.images), -1))
y = digits.target
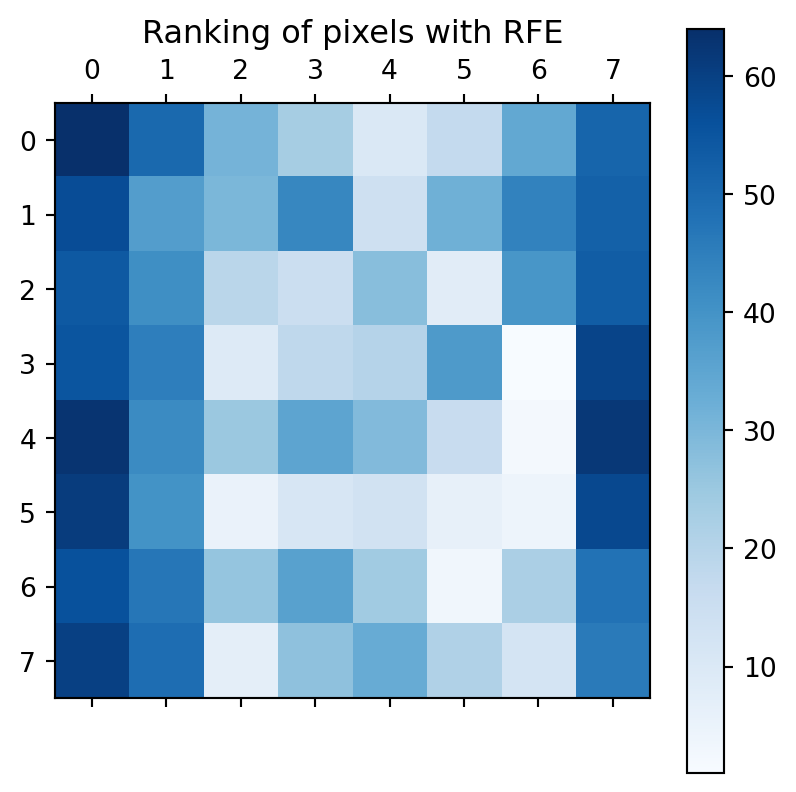
# Create the RFE object and rank each pixel
svc = SVC(kernel="linear", C=1)
rfe = RFE(estimator=svc, n_features_to_select=1, step=1)
rfe.fit(X, y);Vamos a visualizar el ranking por pixel:
Selección de Características con SelectFromModel
SelectFromModel es un meta-transformador que puede usar cualquier estimador que asigne importancia de los atributos:
Si quedan por debajo de un umbral se eliminan.
Se puede usar también
mean,mediany multiplicadores como0.1*mean.
from sklearn.svm import LinearSVC
from sklearn.feature_selection import SelectFromModel
#Use a LinearSVC as the base model for feature selection
lsvc = LinearSVC(C=0.01, penalty="l1", dual=False).fit(X_iris, y_iris)
#Build a model-based selector with the lsvc to reduce the number of features of iris, preserving only thos avobe the average relevance
SFmodel = SelectFromModel(lsvc, prefit=True, threshold="1.25*mean")
X_reduced_model = SFmodel.transform(X_iris)
print(X_iris.iloc[:5,:])
print(X_reduced_model.shape)
print(X_reduced_model[:5,:]) sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
3 4.6 3.1 1.5 0.2
4 5.0 3.6 1.4 0.2
(150, 2)
[[3.5 1.4]
[3. 1.4]
[3.2 1.3]
[3.1 1.5]
[3.6 1.4]]Cambiamos el modelo:
from sklearn.ensemble import ExtraTreesClassifier
#now we do the same but using a tree classifier as the base model for feature selection
clf = ExtraTreesClassifier(n_estimators=50)
clf = clf.fit(X_iris, y_iris)
SFmodel = SelectFromModel(clf, prefit=True, threshold="1.25*mean")
X_reduced = SFmodel.transform(X_iris)
#We can use any model that computes the faeture importance a the base model
print(clf.feature_importances_)
print(X_reduced.shape)[0.08416323 0.05199978 0.41812623 0.44571076]
(150, 2)#We can do the same for the diabetes dataset
diabetes = datasets.load_diabetes()
X_diabetes = diabetes.data
y_diabetes = diabetes.target
from sklearn.linear_model import RidgeCV
ridge = RidgeCV(alphas=np.logspace(-6, 6, num=5)).fit(X_diabetes, y_diabetes)
importance = np.abs(ridge.coef_)
df = pd.DataFrame({"feature_names": diabetes.feature_names, "importance": importance})
sns.catplot(x="feature_names", y="importance", data=df, kind="bar",
errorbar=None, aspect=2, height=4, color="skyblue")
plt.show()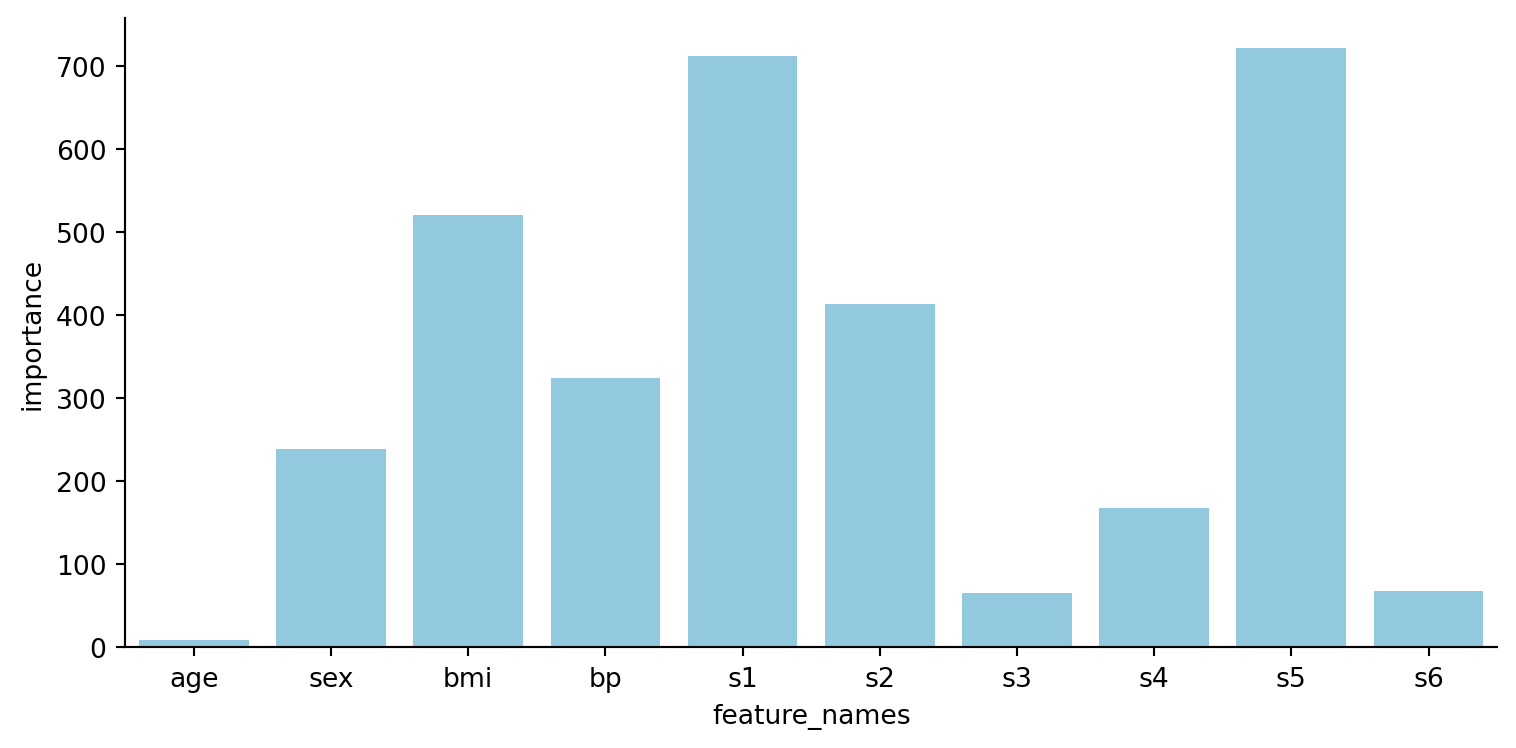
Selección de Características Secuencial
La selección secuencial (Forward-SFS) busca iterativamente una nueva característica a añadir a las ya seleccionadas. Empieza con cero características y escoge aquella que maximiza aplicando CV usando un estimador (cualquiera le vale, pero mejor que no sea lento) sobre una única característica.
Luego repite el procedimiento añadiendo una nueva característica cada vez, hasta terminar con el número pedido de características.
Backward-SFS
Backward-SFS sigue la misma idea, pero al revés, en vez de ir añadiendo va eliminando características aplicando un estimador.
No dan los mismos resultado, ni son igualmente eficientes. Si tenemos 10 características y queremos siete será más eficiente Backward-SFS que Forward-SFS.
Scikit-learn ofrece SequentialFeatureSelector que implementa ambos ( direction puede ser forward o backward).
Ejemplo:
from sklearn.feature_selection import SequentialFeatureSelector
import warnings
warnings.filterwarnings('ignore')
#Perform FORDWARD feature selection over the diabetes dataset to reduce it to 3 dimensions
sfs_forward = SequentialFeatureSelector(clf, n_features_to_select=3, direction="forward")
sfs_forward_fitted = sfs_forward.fit(X_diabetes, y_diabetes)
X_reduced_for = sfs_forward.transform(X_diabetes)
print(X_reduced_for.shape)
print(sfs_forward_fitted.get_support())
atribs = np.array(diabetes.feature_names)
print("Atributos elegidos")
print(atribs[sfs_forward_fitted.get_support()])(442, 3)
[False True False True False False False True False False]
Atributos elegidos
['sex' 'bp' 's4']Aplicando con backward
#Perform BACKWARD feature selection over the diabetes dataset to reduce it to 3 dimensions
sfs_backward = SequentialFeatureSelector(clf, n_features_to_select=3, direction="backward")
sfs_backward_fitted = sfs_backward.fit(X_diabetes, y_diabetes)
X_reduced_back = sfs_backward_fitted.transform(X_diabetes)
print(X_reduced_back.shape)
print(sfs_backward_fitted.get_support())
print("Atributos elegidos")
print(atribs[sfs_backward_fitted.get_support()])(442, 3)
[False False True False True False True False False False]
Atributos elegidos
['bmi' 's1' 's3']Desbalanceo y Reducción de Instancias
Reducción de Instancias (Under-Sampling)
A menudo los datos contienen instancias redundantes. Como el tamaño puede afectar a la calidad de los resultados (y el tiempo) vamos a intentar reducirlo.
También es importante cuando las muestras están poco balanceadas.
Veremos dos enfoques: generación de prototipos, y selección de prototipos.
Usaremos en este apartado el estupendo paquete imblearn, documentado en https://imbalanced-learn.org/stable/
Generación de Prototipos
En este caso se reducen las muestras pero las que quedan son generadas (no seleccionadas) del original. La técnica más usada es el K-Means, y sintetizar cada clase con un centroide del K-Means en vez de uar las muestras originales.
Vamos a crear unos datos de ejemplo:
from sklearn.datasets import make_classification
from collections import Counter
#Create an artificial datasets to see the effects of under-sampling and over-sampling
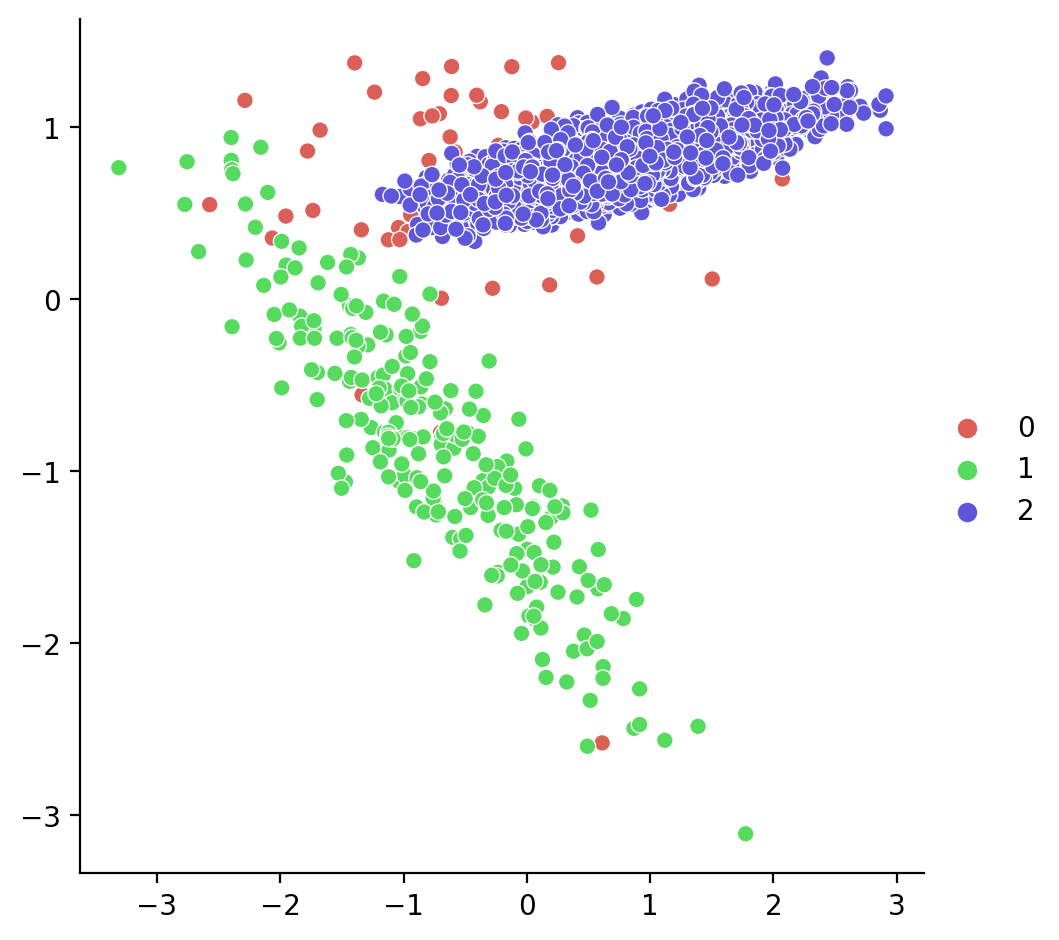
X, y = make_classification(n_samples=5000, n_features=2, n_informative=2,
n_redundant=0, n_repeated=0, n_classes=3,
n_clusters_per_class=1,
weights=[0.01, 0.05, 0.94],
class_sep=0.8, random_state=10)
print(sorted(Counter(y).items()))[(0, 67), (1, 264), (2, 4669)]Están poco balanceadas.
Visualizamos los puntos:
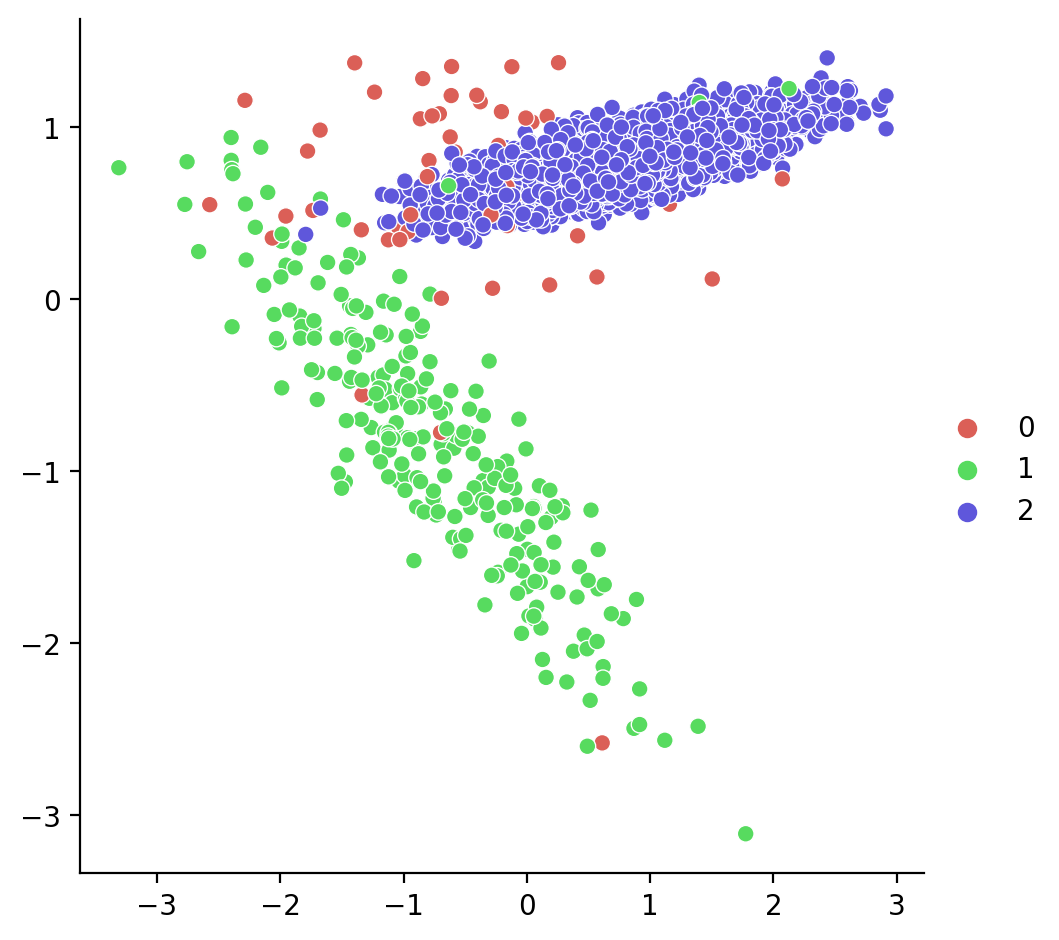
Aplicamos el under_sampling:
from imblearn.under_sampling import ClusterCentroids
#Perform clustering-based prototype generation
cc = ClusterCentroids(random_state=0, voting="soft")
X_resampled, y_resampled = cc.fit_resample(X, y)
print(sorted(Counter(y_resampled).items()))
reduced = (X.shape[0]-X_resampled.shape[0])/X.shape[0]
print(f"Reduce el {100*reduced} %")[(0, 67), (1, 67), (2, 67)]
Reduce el 95.98 %Ahora cada clase está balanceada.
Visualizamos la salida:
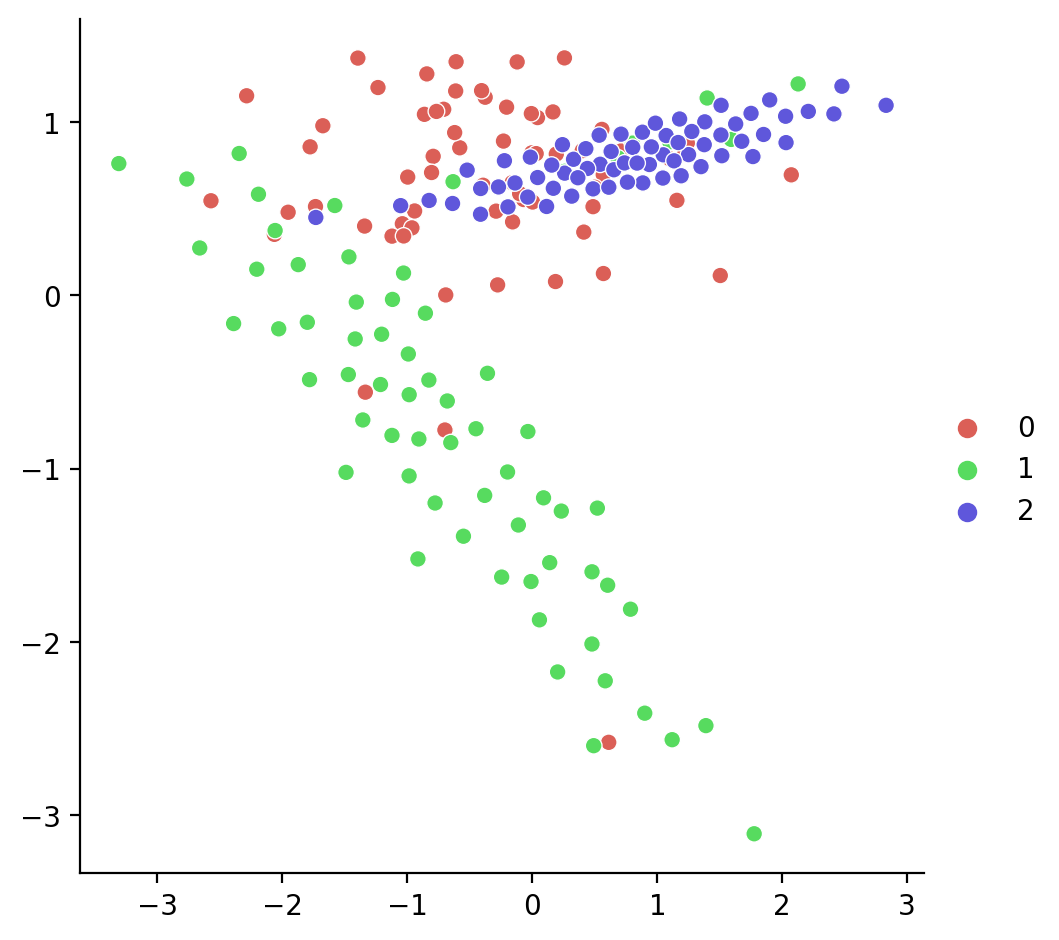
Selección de prototipos
En este enfoque se muestrean menos instancias pero las que quedan son instancias del conjunto original.
En imblearn hay muchos, escogeremos dos:
- Aleatorio: Random under sampling technique, una técnica rápida muestreando aleatoriamente para cada clase.
- Usando vecindario: Edited nearest neighbours, usa el vecindario para eliminar instancias demasiado alejadas del vecindario.
Random under sampling technique
from imblearn.under_sampling import RandomUnderSampler
#Perform random prototype selection
rus = RandomUnderSampler(random_state=0)
X_resampled, y_resampled = rus.fit_resample(X, y)
print(sorted(Counter(y_resampled).items()))
reduced = (X.shape[0]-X_resampled.shape[0])/X.shape[0]
print(f"Reduce el {100*reduced:.2} %")[(0, 67), (1, 67), (2, 67)]
Reduce el 9.6e+01 %Vamos a mostrarlo:
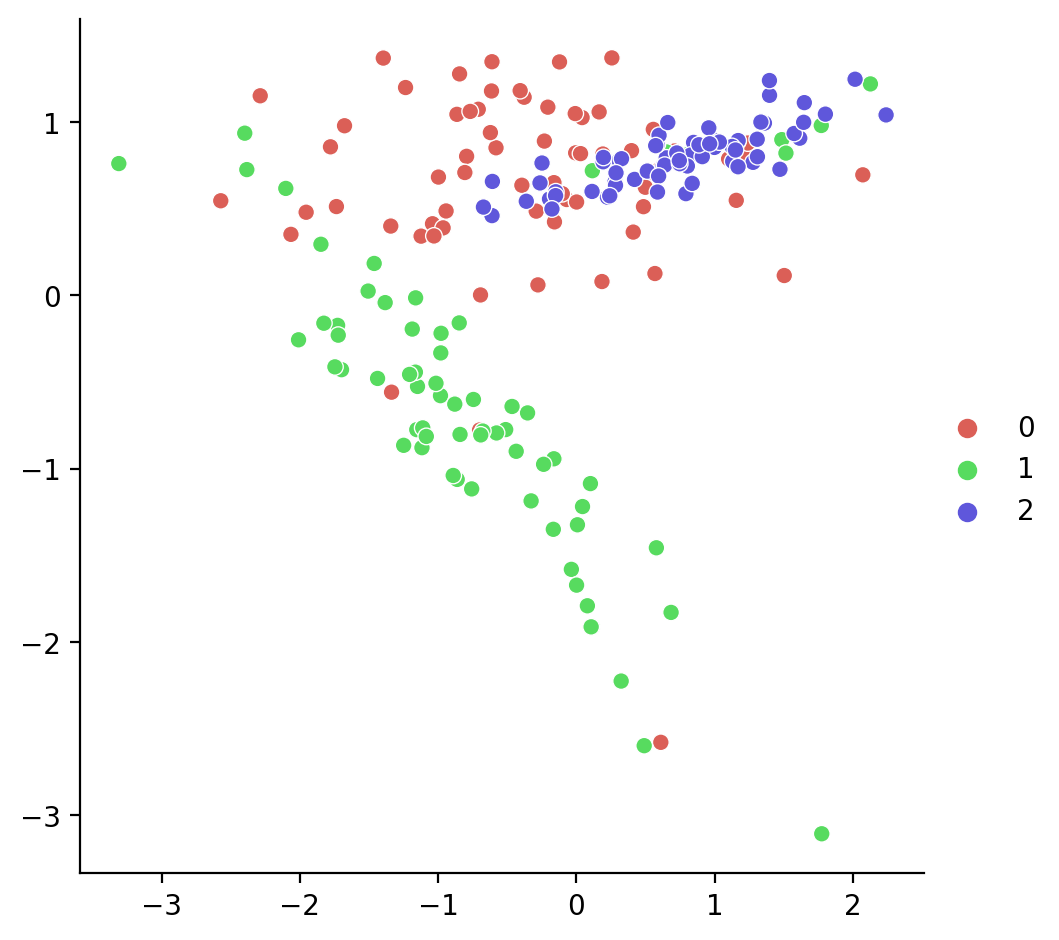
Edited Nearest Neighbours
Edited Nearest Neighbours aplica el algoritmo de vecinos más cercanos y borra las instancias que no son suficientemente similares a las del vecindario.
Para cada instancia, se calculas sus vecinas y si no se cumple el criterio se borra.
Existen dos criterio a la hora de comparar las vecindas:
La mayoría (
kind_sel='mode') deben pertenecer a la misma clase.Que todas (
kind_sel='all') pertenezcan a la misma clase.
El primero es más conservador y el segundo excluye más.
Aplicamos primero el más conservador:
from imblearn.under_sampling import EditedNearestNeighbours
enn = EditedNearestNeighbours(kind_sel="all")
X_resampled, y_resampled = enn.fit_resample(X, y)
print(X_resampled.shape)
print(sorted(Counter(y_resampled).items()))
reduced = (X.shape[0]-X_resampled.shape[0])/X.shape[0]
print(f"Reduce el {100*reduced:.2} %")(4824, 2)
[(0, 67), (1, 223), (2, 4534)]
Reduce el 3.5 %Visualizamos:
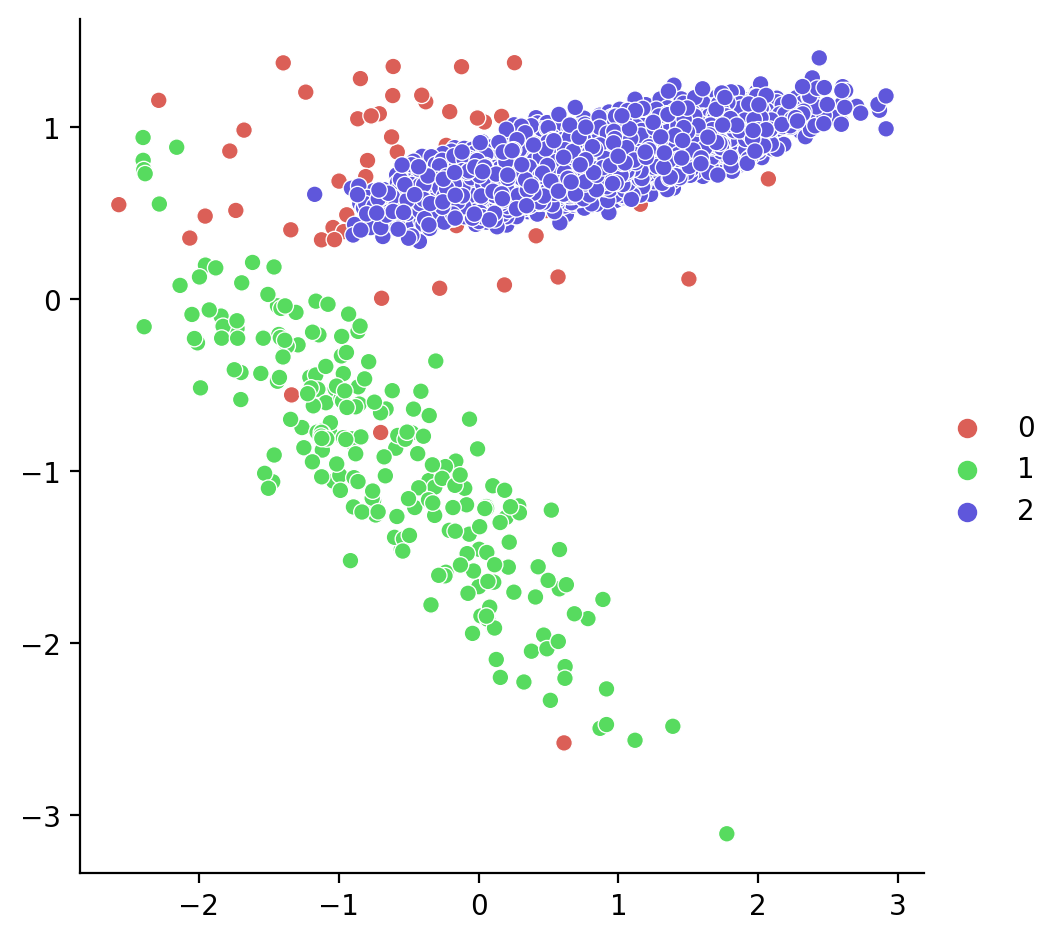
Aplicamos el menos conservador:
from imblearn.under_sampling import EditedNearestNeighbours
enn = EditedNearestNeighbours(kind_sel="mode")
X_resampled, y_resampled = enn.fit_resample(X, y)
print(X_resampled.shape)
print(sorted(Counter(y_resampled).items()))
reduced = (X.shape[0]-X_resampled.shape[0])/X.shape[0]
print(f"Reduce el {100*reduced:.2} %")(4972, 2)
[(0, 67), (1, 244), (2, 4661)]
Reduce el 0.56 %Visualizamos:
Sobre Muestreo
Aunque no es reducción vamos a ver cómo se pueden sobre-muestrear las instancias para tener más ejemplares de las clases minoritarias y obtener mejor resultados.
imblearn ofrece varios, como el aleatorio, SMOTE, entre otros. Vamos a ver los dos primeros.
Primero creamos los datos
from sklearn.datasets import make_classification
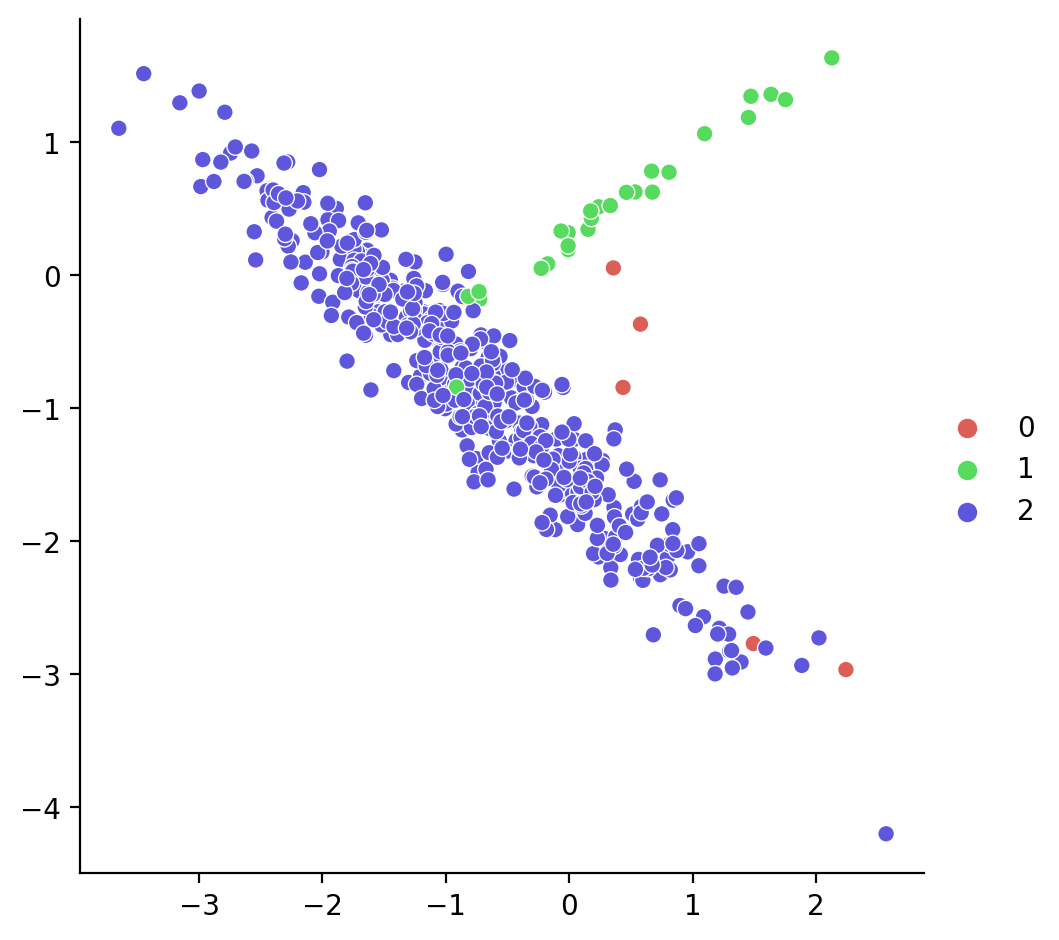
X, y = make_classification(n_samples=500, n_features=2, n_informative=2,
n_redundant=0, n_repeated=0, n_classes=3,
n_clusters_per_class=1,
weights=[0.01, 0.05, 0.94],
class_sep=0.8, random_state=42)
print(sorted(Counter(y).items()))[(0, 5), (1, 27), (2, 468)]Vamos a visualizar el original:
RandomOverSampler
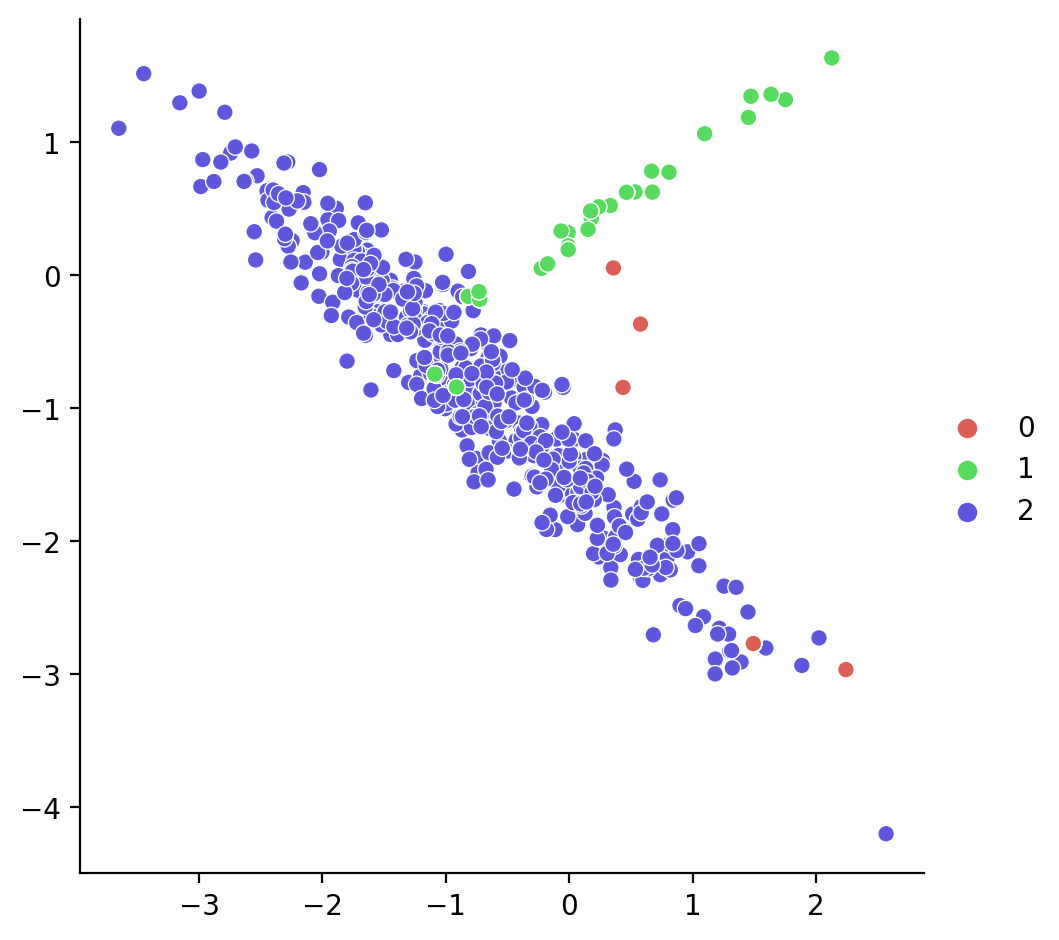
Ahora aplicamos el RandomOverSampler
Vamos a aplicarlo:
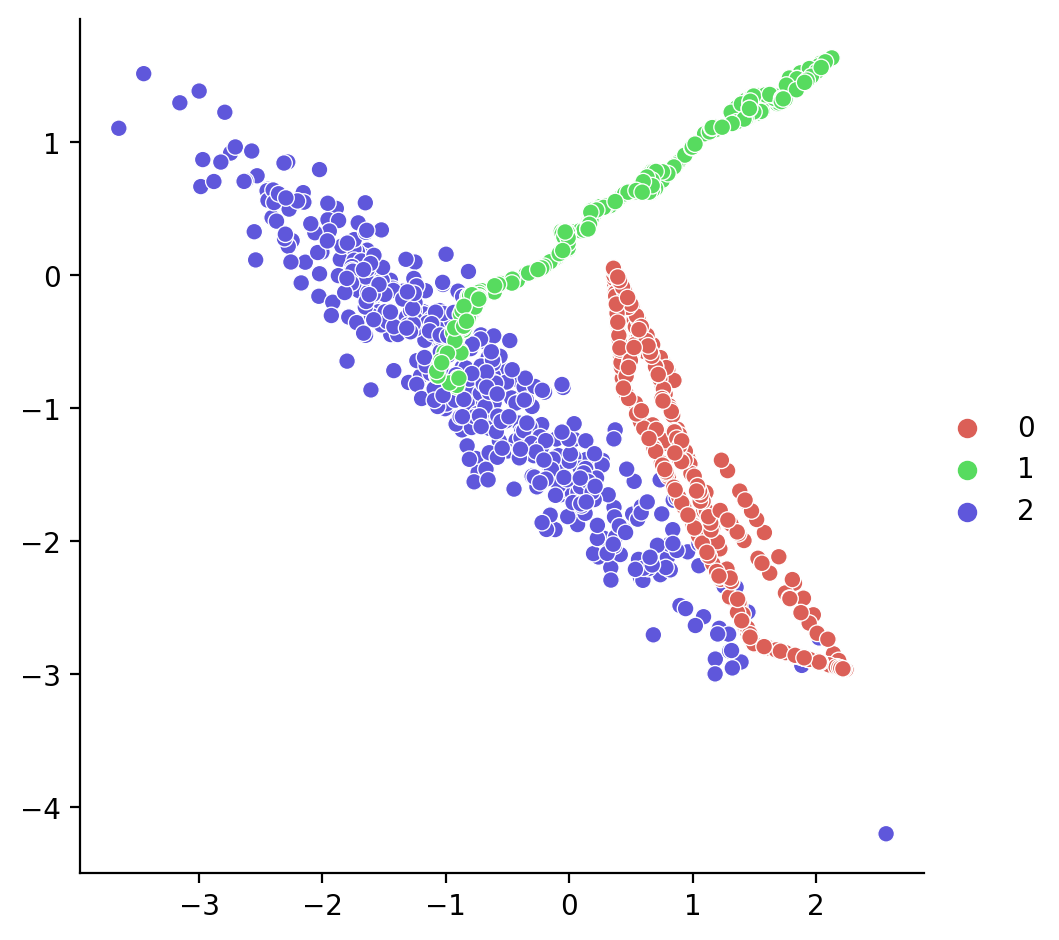
SMOTE
Otra técnica clásica es SMOTE, que genera datos de forma sintética.
Vamos a aplicarlo:
Datos anómalos
Datos anómalos
A veces en los datos se presentan valores anómalos, que se han introducido, por ejemplo, debido a errores en los procesos de recogida de datos.
Quizás el valor anómalo se deba a una cambio en la distribución de valores y no a un error.
La intuición básica en las técnicas detección de anomalías es:
- La mayoría de los datos siguen una determinada distribución.
- Los datos las anomalías representan entonces una distribución distinta, que no coincide con el resto.
Detección a mano
Un método clásico es considerar como datos anómalos aquellos para los que el valor de un atributo esté fuera del 1.5*rango intercuartil
Supongamos unos datos:
# Generate train data
rng = np.random.RandomState(42)
X_orig = 0.3 * rng.randn(100, 2)
#X_good = X_orig-4
X_good = np.r_[X_orig + 2, X_orig - 2]
# Generate some abnormal novel observations
X_outliers = rng.uniform(low=5, high=8, size=(10, 2))
X = np.vstack([X_good, X_outliers])
np.random.shuffle(X)
X_df = pd.DataFrame(X, columns=["V1", "V2"])
print(X_df.shape)
print(X_df.columns)(210, 2)
Index(['V1', 'V2'], dtype='object')
Vamos a aplicar:
Visualizamos:
Detección Outliers usando scikit-learn
Scikit-learn ofrece distintos algoritmos para detectar outliers En la Documentación

Vamos a probar Isolation Forest.
Isolation Forest
‘Aisla’ observaciones aleatoriamente escogiendo una característica y aleatoriamente divide recursivamente según sus valores, usando una estructura de árbol.
Cerca de -1 si lo considera outlier, 1 en caso contrario.
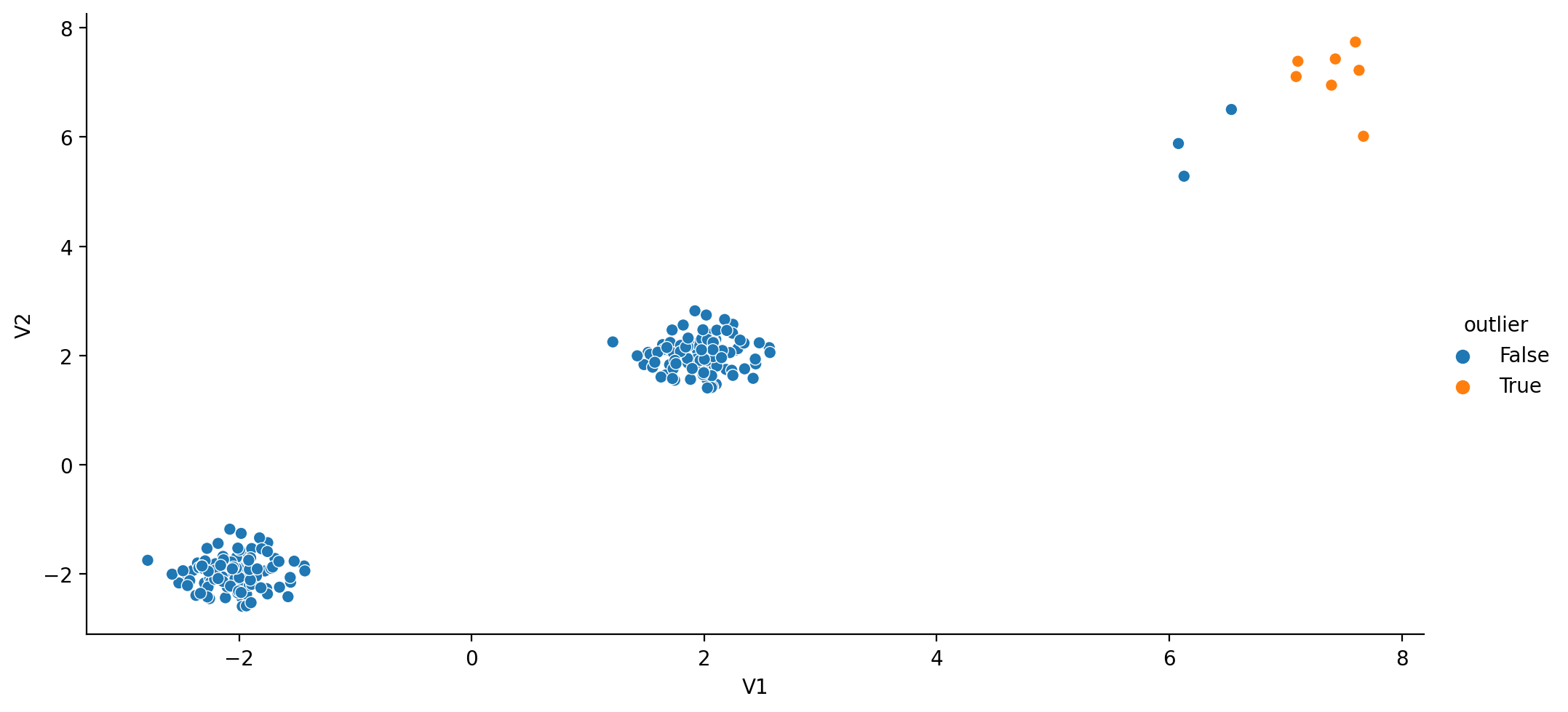
from sklearn.ensemble import IsolationForest
clf = IsolationForest(n_estimators=10, warm_start=True)
clf.fit(X_df) # fit 10 trees
X_df_if = X_df.copy()
X_df_if["outlier"] = clf.predict(X_df) < 1
print(X_df_if.head(3)) V1 V2 outlier
0 1.819488 2.555683 True
1 -1.753382 -1.430962 True
2 1.836685 2.033277 FalseVisualizamos:
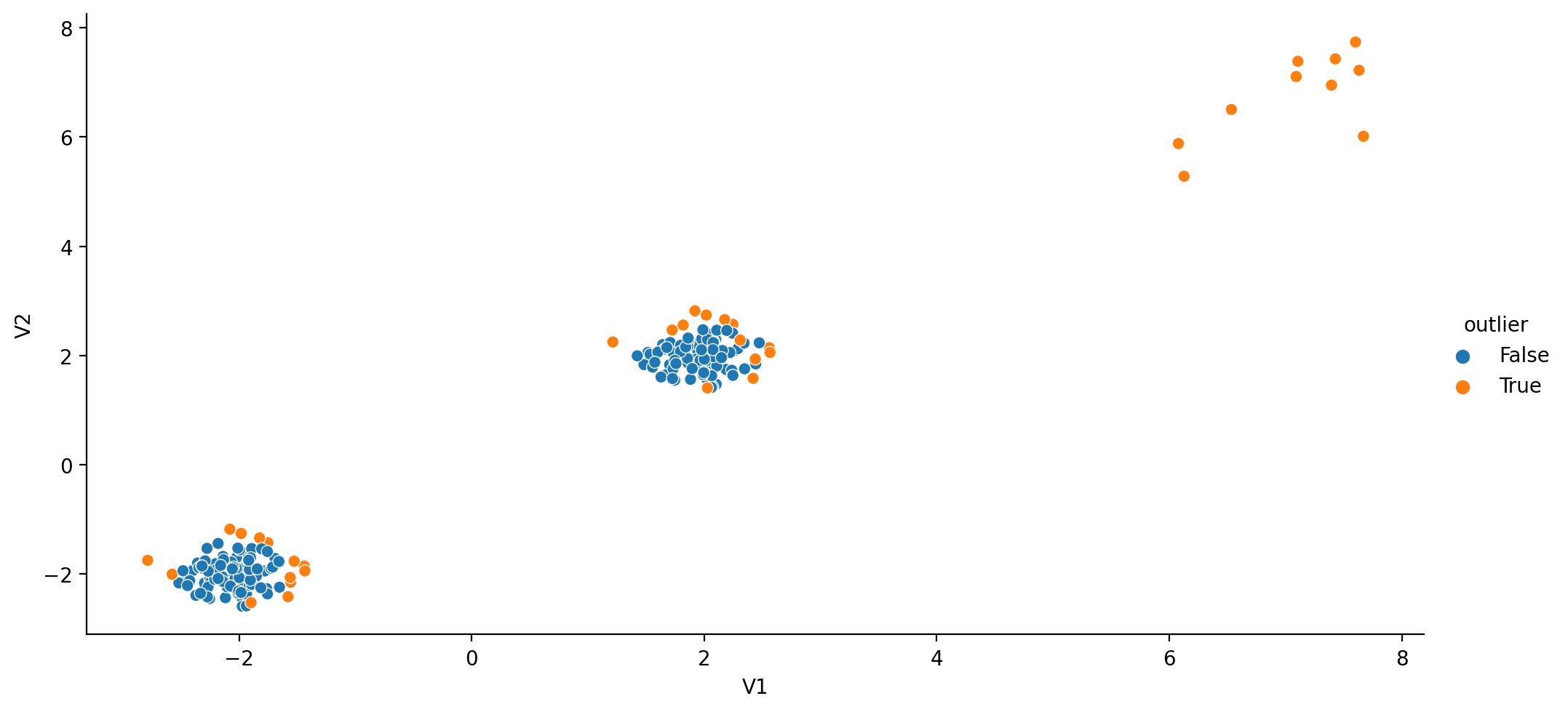
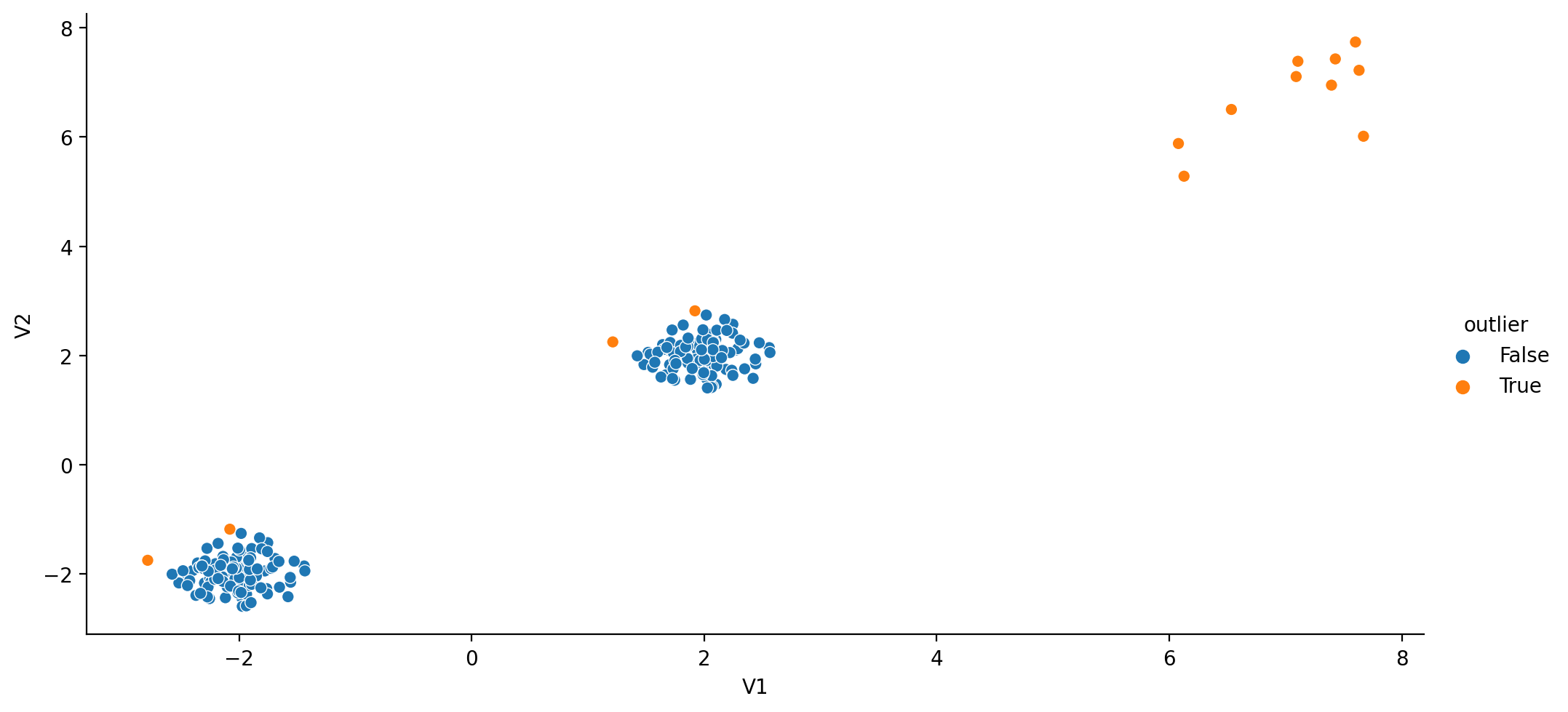
LocalOutlierFactor
Esta técnica mide la desviación local de una muestra respecto a los vecinos (usando k-vecinos). Al comparar la distancia local con la de los vecinos, se ientifica las instancias con una densidad sustanciamente menor que sus vecinos.
Cerca de -1 si lo considera outlier, 1 en caso contrario.
Visualizamos:
Ruido y Filtros
Ruido y Filtros
En muchos casos existe ruido o inexactitudes en los parámetros.
Hay que evitar el sobre-aprendizaje que puede llegar a aprender dicho ruido.
Tiene especial interés cuando hay una variable que cambia con el tiempo, hay muchos filtros asociado a series temporales.
En R existe el paquete NoiseFiltersR pero no tiene contrapartida similar en Python.
Veremos filtros disponibles en scipy.signal.
Señales temporales
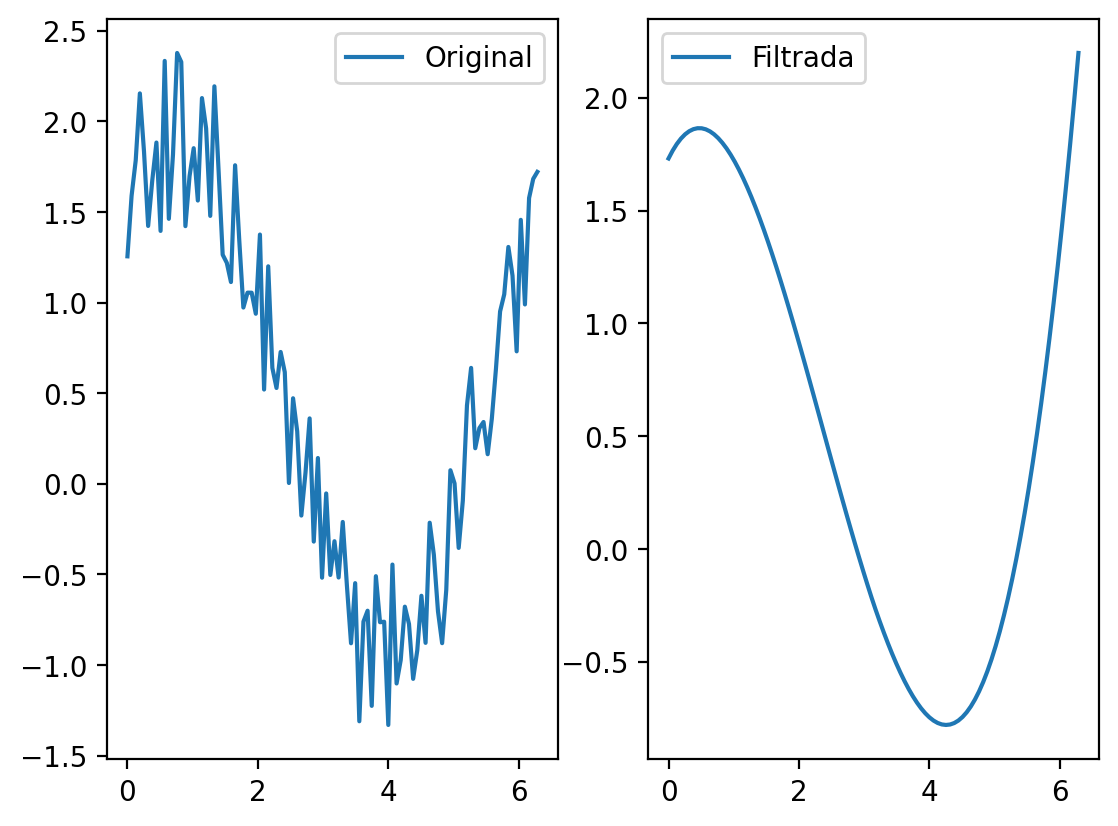
Aplicamos el filtro Savitzky-Golay
Imágenes
En las imágenes es muy común que exista ruido, vamos a ver varios filtros que los eliminan.
Para ello usaremos el paquete scipy y opencv-python (muy conocida).
Se puede instalar con:
import cv2
fig, axs = plt.subplots(1,2)
image_orig=cv2.imread("squirrel_cls.jpg", cv2.IMREAD_GRAYSCALE)
size=(image_orig.shape[0],image_orig.shape[1])
white_noise = np.random.randint(-20, 40, size = image_orig.shape)
image=image_orig+white_noise
image=np.maximum(np.zeros(size), image)
image=np.minimum(255*np.ones(size), image)
axs[0].imshow(image_orig, cmap='gray'); axs[0].set_title("Original")
axs[1].imshow(image, cmap='gray'); axs[1].set_title("Con Ruido")
plt.show()
Vamos a usar wiener

Comparamos cómo queda respecto al original

Aplicamos cv2.fastNlMeansDenoising

Comparamos los resultados de los dos filtrados

Ejercicios
Ejercicios de dimensionalidad
Vamos a usar el datasets de los pingüinos:
Reduce usando PCA o el cluster agglomerativo la dimensionalidad de los valores numéricos del problema en 2 componentes. Nota: Es cómodo usar el atributo select_dtypes de pandas para filtrar atributos de tipo np.number.
Visualiza la representación, resaltando la especie. ¿Es separable en dos?
Ejercicios de selección de características
Vamos a usar aquí el dataset de estudiantes.
Target es el valor con el que se quiere predecir, que indica si los estudiantes terminan la carrera o no.
Elimina directamente los atributos con valores perdidos, y usa StandardScaler sobre el resto.
Separa el atributo Target en otra variable.
Visualizar usando
yellobricksla importancia de las características.Carga el estimador
Usando SelectModel con ese estimador eliminar el 20% de los atributos.
Usa SequentialFeatureSelector con ese estimador para eliminar el 20% de los atributos en ambas direcciones, ¿cuáles son eliminados?
¿Son iguales en ambas direcciones?
Ejercicio de Balanceo
Students no está bien balanceado. Haz un Random Under sampling y comprueba que ahora sí esté balanceado.
Haz un Edited Nearest Neighbours sobre el original menos conservador, ¿cuántos ha quitado?
Ejercicio de anomalías
- Elimina Datos anómalos usando LocalOutlierFactor e indica el porcentaje de eliminados.